Servicios Personalizados
Revista
Articulo
Links relacionados
Compartir
Agrociencia (Uruguay)
versión impresa ISSN 1510-0839versión On-line ISSN 2301-1548
Agrociencia Uruguay vol.17 no.1 Montevideo jun. 2013
Desarrollo de un multiplex de microsatélites para diagnóstico de paternidad en ovinos Corriedale del Uruguay
Peraza Pablo1, Rincón Gonzalo2, Ravagnolo Olga3, Dalla Rizza Marco1, Kelly Lucy1
1Unidad de Biotecnología Animal, Instituto Nacional de Investigación Agropecuaria. Ruta 48 km 10, Rincón del Colorado, Canelones, Uruguay. Correo electrónico: pperaza@inia.org.uy.
2Animal Science Department, Universidad de California. Davis, USA.
3Programa de Carne y Lana. Instituto Nacional de Investigación Agropecuaria.
Recibido: 30/3/12 Aceptado: 17/12/12
Resumen
En el Uruguay la raza Corriedale representa el 70% del stock ovino, por lo cual es importante garantizar la exactitud al realizar un diagnóstico de paternidades mediante tipificación de ADN, con el fin de asistir a los registros genealógicos y a los programas de selección para las características de importancia económica realizados en dicha raza. En el presente trabajo se desarrolló un multiplex de 10 microsatélites (Short Tandem Repeats, STR) recomendados del panel de la ISAG (International Society of Animal Genetics). Con este multiplex se analizó una población de 156 ovinos de la raza Corriedale del Uruguay. El objetivo del presente trabajo fue evaluar dicho multiplex de acuerdo a variabilidad genética de las frecuencias alélicas para la asignación de progenitores mediante los valores de su Probabilidad de Exclusión (PE). Los STR presentaron una variabilidad relativamente alta de acuerdo al Índice de Heterocigosidad promedio (HObs=0,661), al número de alelos observados (N=8,6) y al Contenido de Información Polimórfica (PIC=0,614). Se observó en cuatro de los 10 STR utilizados (FCB20, TGLA53, MCM527 y MCM130) una pérdida de equilibrio Hardy-Weinberg debido a la disminución significativa de heterocigotos, con la consiguiente reducción de variabilidad genética. Como consecuencia la Probabilidad de Exclusión combinada en dicha población presentó un valor de 0,960. Concluimos que para mejorar la certeza en el diagnóstico de paternidad se podría incluir un mayor número de STR en el multiplex, dada la baja variabilidad genética presente en los padres.
Palabras clave: TIPIFICACIÓN ADN, OVINOS, STR
Summary
Development of a Microsatellite Multiplex for Paternity Testing in Uruguayan Corriedale Sheep
The Corriedale breed represents 70% of Uruguayan sheep stock, therefore it is important to guarantee accuracy in paternity assessment through DNA analysis, to contribute to genealogy recording and selection programs for economically relevant traits of the breed. In this study, a multiplex of 10 microsatellites (Short Tandem Repeats, STR) recommended from the ISAG (International Society of Animal Genetics) panel was developed. With this multiplex, a population of 156 Corriedale sheep was analyzed. The aim of this study was to evaluate the multiplex according to genetic variability of allele frequencies, for assigning parents through the their Probability of Exclusion (PE) values. The STR showed a relatively high variability according to the average Heterozygosity Index (HObs=0.661), the number of alleles observed (N=8.6), and the Polymorphic Information Content (PIC=0.614). In four ot the 10 STR (FCB20, TGLA53, MCM527 and MCM130) there was a loss of Hardy-Weinberg equilibrium due to the significant decrease of heterozygotes, with a consequent reduction of genetic variability. As a result, combined exclusion probability presented a value of 0.960. We conclude that in order to improve the accuracy in paternity diagnostic the multiplex could include a greater number of STR, given the low genetic variability present in the parents.
Key words: DNA ANALYSIS, SHEEP, STR
Introducción
La raza Corriedale se originó en Nueva Zelanda por el cruzamiento de la raza Lincoln y Merino e ingresó al Uruguay en 1916. Se utiliza en nuestro país con el doble propósito de producir carne y lana, constituyendo el 70% del stock ovino uruguayo (Cardellino et al., 1994). En la actualidad se llevan a cabo evaluaciones genéticas a partir de datos disponibles desde del año 1969, en que se comenzaron a utilizar las planillas de «Flock Testing». Se cuenta con datos de 11 características productivas en esta raza, obteniéndose DEPs (Diferencia Esperada en Progenie) que permiten evaluar objetivamente ovinos reproductores por sus características de valor productivo en planteles denominados como puro de origen o de pedigree. Las características medibles evaluadas son: peso vivo al destete, recría, esquila, lana en la cara, score de pigmentación, peso de vellón sucio, rendimiento al lavado (vellón limpio), coeficiente de variación del diámetro de fibra, largo de mecha y resistencia a parásitos gastrointestinales mediante el conteo de huevos por gramo de materia fecal (HPG) a diferentes edades (Ciappesoni et al., 2012).
Se debe tener en cuenta que la asignación incorrecta de paternidades puede tener efecto en la tasa de ganancia genética (Israel y Weller, 2000). En este sentido, se debe considerar de importancia la realización de diagnósticos de paternidades ya que la asignación de la descendencia puede no ser correcta debido al intercambio madre-cordero durante las pariciones. Por otro lado, cuando la encarnerada de la majada general se realiza mediante servicios múltiples con carneros de alto valor de cría para diferentes características mejoradoras de la especie, el número de descendientes va a depender de las relaciones de dominancia-subordinación de los carneros utilizados (Ungerfeld y González-Pensado, 2008; Tilbrook et al., 1987).
Desde 1989 los microsatélites (Short Tandem Repeat: STR) son los marcadores elegidos para estudios a nivel genómico en varios organismos por ser co-dominantes y poseer una alta tasa de mutación (Weber y May, 1989). Esto se refleja en su alto índice polimórfico, fácil genotipado mediante técnicas moleculares (Kelly y Postiglioni, 1997), y en el hecho de que estén ampliamente distribuidos en el genoma ovino (Maddox et al., 2001; Beraldi et al., 2006). También es importante considerar que la mayoría de los microsatélites utilizados en este trabajo son avalados por la Sociedad Internacional de Genética Animal (ISAG). Allí se realiza bianualmente una estandarización de resultados entre varios laboratorios participantes.
Nuestro objetivo por lo tanto fue desarrollar y evaluar un multiplex utilizando marcadores microsatélites con el propósito de determinar con alta probabilidad las relaciones de paternidad para la raza Corriedale. Esta herramienta, brindará información relevante a los programas de selección y mejoramiento genético ovino.
Materiales y métodos
En el presente estudio se realizó un servicio múltiple con seis carneros de pedigree en una majada de madres de raza Corriedale general. Al cabo del periodo gestacional se obtuvo una población de 150 corderos. Se les extrajo una muestra de sangre por punción venosa mediante la utilización de un anticoagulante K2EDTA (AMRESCO®). La extracción de ADN se realizó con DNAzol® (GIBCO Life Technologies) según protocolo de fabricante y se almacenó a temperatura ambiente en 100ul de NaOH 8 mM. Se evaluó concentración por absorción a 260/280 nm y 230/260 nm en un equipo Thermo Scientific NanoDrop 8000 verificando buena cantidad y calidad de material. Para el presente trabajo se utilizaron los siguientes STR: MCM214, MCM527, CSRD247, D5S2, OarFCB20, OarFCB340, TGLA53, BM1818, MCM130, CSRD2138 (Cuadro 1). Los mismos fueron seleccionados por varios factores: el largo de los fragmentos amplificados para adjudicarle diferentes fluorocromos (TAMRA, FAM, HEX) e interpretar el electroferograma; el fluorocromo utilizado en cada microsatélite; localización cromosómica (GeneBank); recomendación del panel de STR del International Comparison Test - ISAG y su compatibilidad de amplificación en la reacción en cadena de la polimerasa (PCR) dada por la temperatura de hibridación de cada primer. El multiplex desarrollado por INIA (DNA-OV. Biotec) está formado por dos paneles de STR: el panel 1 compuesto por los microsatélites MCM214, MCM357, CSRD247, D5S2, OarFCB20, OarFCB34 TGLA53 y el panel 2 con los microsatélites BM1818, MCM130 y CSRD2138.
Se amplificaron estos STR por PCR ajustando su temperatura de hibridación según la mejor resolución de los amplicones. Las amplificaciones fueron realizadas en un termociclador Corbett Research, modelo: CG1-96. El genotipado se realizó en un secuenciador ABI PRISM 310 Genetic Analyzer, Applied Biosystems. El análisis de los fragmentos se realizó con el programa Genotyper 3.1 Applied Biosystems. Heterocigosidad observada (HObs), Frecuencias Alélicas (FA), Contenido de la Información Polimórfica (PIC), significancia de la desviación del equilibrio Hardy-Weinberg (HW) y Probabilidad de Exclusión (PE) se calcularon utilizando el programa CERVUS 2.0 (Kalinowski et al., 2007). Los cálculos para desequilibrio de ligamiento se realizaron con el programa GENEPOP, versión 3.1b (Raymond y Rousset, 1995; Rousset, 2008).
Resultados y discusión
Empleando el kit multiplex desarrollado por INIA (DNA-OV. Biotec) se logró obtener un perfil de amplificación de los animales que permitió su posterior análisis. En la categoría corderos, se obtuvieron entre ocho y 13 alelos por marcador, indicando un número mayor al encontrado en la categoría carneros presentando entre dos y cinco alelos.
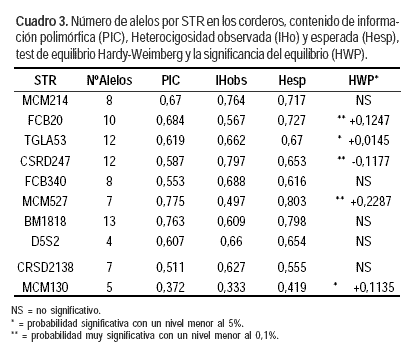
El Índice de Heterocigosidad observada promedio (HObs) de los STR presentó un valor de 0,661 y la PE de 0,960 (Cuadro 2). La mayor Heterocigocidad observada individual fue de 0,785 para el STR CSRD247 y la menor fue para el STR MCM527 de 0,490. Sin embargo en este último STR su PIC fue el más alto 0,773.
De acuerdo al estudio realizado en la raza Corriedale por Tomasco et al. (2002) con 10 STR diferentes a los empleados en el presente trabajo y con 77 animales de la misma raza se observó un HObs ligeramente mayor (0,710). Esto puede deberse probablemente a que los animales pertenecían a diferentes establecimientos como también a la no existencia, como en nuestro estudio, del posible caso de un carnero dominante en la majada.
Como se aprecia en el Cuadro 2, el valor de la desviación del equilibrio Hardy-Weinberg fue significativo para cuatro STR (FCB20, CSDR247, MCM527 y TGLA53). Una de las causas podría ser la selección producida por los seis carneros padres aportando pocos alelos con alta frecuencia y a la existencia de un carnero dominante. Esto se detectó a la hora de asignar las relaciones de parentesco, determinando que el 43% de la descendencia corresponde a un solo carnero.
El STR CSRD247 fue el único que presentó un aumento muy significativo de heterocigotos observados, probablemente por haber influido el efecto migratorio que depende del número de individuos que migran y la diferencia de las frecuencias génicas entre las dos sub poblaciones (madres pertenecientes a un rodeo Corriedale general y carneros de otra cabaña de pedigree) (Kalinowski et al., 2007). Al formarse la majada de corderos a partir de dos sub poblaciones con frecuencias génicas diferentes y al ingreso de un carnero dominante, se observa una baja en la heterocigosis de determinados marcadores STR. Además, CSRD247 en los corderos presentó alta frecuencia del genotipo del padre dominante (genotipo CSRD247: 231/233) que dejó el 43% de los corderos, pero en la majada general los corderos a su vez tienen tres alelos (217, 241, 243) con frecuencias intermedias (0,101, 0,023 y 0,017: Cuadro 3) probablemente aportados por las madres. En el Cuadro 3 se puede observar también un aumento en la frecuencia alélica de entre dos y tres alelos por marcador. Estos, por lo general, concuerdan con los del carnero dominante, aunque la variación en las frecuencias alélicas entre este carnero y el resto no representaron grandes variaciones. En este caso se observa el efecto de la migración poblacional que está determinada por el número de individuos inmigrantes (carneros) y de las diferencias entre las frecuencias génicas produciendo en las crías un efecto de aumento de heterocigosidad debido a la diferencia de estas frecuencias génicas entre nativos (madres) y migrantes (padres) (Cardellino y Rovira, 1987).
Finalmente, se asignó un único padre al 83% de los hijos debido a que el panel de 10 STR presentó una HObs medio de 0,661 y una PE de 0,960. Si bien la tipificación de ADN mediante la utilización de marcadores moleculares es una herramienta poderosa para el productor agropecuario, ya que le permite diagnosticar paternidades de manera precisa e inequívoca dentro de un rebaño, este va a depender del polimorfismo de los microsatélites seleccionados. Más aún en casos como este, donde nos encontramos con que no hay gran variabilidad en la población con estos marcadores. Respecto a este último punto es recomendable analizar los STR en los padres antes de realizar servicios múltiples, permitiendo así seleccionarlos tomando los más polimórficos para cada caso y/o anexando más cantidad de STR para aumentar la PE con el fin de asignar el 100% de paternidades. De esta forma la tipificación de ADN con STR permite corroborar las genealogías de los animales indistintamente del tipo de manejo reproductivo.
El Kit DNA-Ov.Biotec presenta dos STR (CSRD2138 y MCM527) que se localizan en el cromosoma 5 (Cuadro 1), y a los cuales se analizó su ubicación en el Sheep MAP V.4.7, localizándose a 38,8cM y 118,9 cM respectivamente. Utilizando el programa GENEPOP V3.1b se calculó el desequilibrio de ligamiento con un número de interacciones de 1000, resultando que no estaban ligados con un p-valor de 0,197 (SE= 0,026) por lo cual segregaban independientemente. De esta forma comprobamos que no se altera su polimorfismo pues no se trasmiten como haplotipos.
Es también importante considerar que el DNA-OV.Biotec presenta dos STR (CRDS2138 y MCM214) ubicados en regiones genómicas vinculadas a la resistencia a parásitos gastrointestinales de acuerdo a varios autores (Benavides et al., 2002; Dominik, 2005; Beh et al., 2002) y a la base de QTL de la NAGRP (National Animal Genome Research Program). Estos STR están vinculados a la resistencia a parásitos gastrointestinales como el Haemoncus contortus y Trichostrongylus columbriformis (Ciappesoni et al., 2012; Benavidez et al., 2002). Es de resaltar además que es una de las características que se evalúan para calcular el mérito genético (DEP-HPG). Actualmente se están estudiando los genes candidatos a esas características de interés productivo con el fin de que asistan a la selección para la resistencia a parásitos gastrointestinales.
Conclusiones
Los resultados obtenidos nos indican que la PE del kit DNA-Ov.Biotec debe ser mayor. Para aumentar su efectividad se anexarán nuevos STR con el fin de apoyar los programas de mejora genética en Corriedale Uruguayo, donde una alta certeza en la identificación del parentesco es fundamental.
Estos estudios también son de gran utilidad para determinar la ganancia en la heterocigosidad en razas productivas que están sometidas a las fuerzas que alteran el equilibrio de Hardy-Weinberg como lo son la migración y (mayoritariamente en nuestros sistemas de mejoramiento) la selección.
Agradecimientos
Universidad de la República, Facultad de Veterinaria.
Bibliografía
Adelson DL, Cam GR, DeSilva U, Franklin IR. 2004.Gene expression in sheep skin and wool (hair). Genomics, 83(1): 95-105.
Beh KJ, Hulme DJ, Callaghan MJ, Leish Z, Lenane I, Windon RG, Maddox JF. 2002 A genome scan for quantitative trait loci affecting resistance to Trichostrongylus colubriformis in sheep. Animal Genetics, 33: 97-106.
Benavides MV, Weimer TA, Borba MFS, Berne MEA, Sacco AMS. 2002. Association between microsatellite markers of sheep chromosome 5 and faecal eggs counts. Small Ruminat Research, 46: 97-105.
Beraldi D, McRae AF, Gratten J, Slate J, Visscher PM, Pemberton JM. 2006. Development of a Linkage Map and Mapping of Phenotypic Polymorphisms in a Free-Living Population of Soay Sheep (Ovis aries). Genetics, 173: 1521-1537.
Bishop MD, Kappes SM, Keele JW, Stone RT, Sunden SLF, Hawkins GA, Solinas-Toldo S, Fries R, Grosz MD, Yoo J, Beattie CW. 1994. A genetic linkage map for cattle. Genetics, 136(2): 619-639.
Buchanan FC, Galloway SM, Crawford AM. 1994. Ovine microsatellites at the OarFCB5, OarFCB19, OarFCB20, OarFCB48,OarFCB129 and OarFCB226 loci. Journal of Animal Genetics, 25(1): 60.
Cardellino R, Salgado C, Azzarini M. 1994. La producción ovina y lanera en Uruguay. Producción Ovina, 7: 7-22.
Cardellino R, Rovira J. 1987. Mejoramiento genético animal. Montevideo: Hemisferio Sur. 253p.
Ciappesoni G, Nicolini P, Kelly L, Grasso N, Peraza P, Cabrera A, Goldberg V. 2012. Molecular characterization of parasite resistant/susceptible Uruguayan Merino lambs. Archivos Latinoamericanos de Producción Animal, 20(1-2): 34 - 41.
Crawford AM, Dodds KG, Ede AJ, Pierson CA, Montgomery GW, Garmonsway HG, Beattie AE, Davies K, Maddox JF, Kappes SW. 1995. An autosomal genetic linkage map of the sheep genome. Genetics, 140(2): 703-724.
Davies KP, Maddox JF, Harrison B, Drinkwater R. 1996. Ovine dinucleotide repeat polymorphism at eight anonymous loci. Animal Genetics, 27: 381-82.
Dominik S. 2005. Quantitative trait loci for internal nematode resistence in sheep : a review. Genetics Selection Evolution, 37(S1): S83-S96
Drinkwater R, Harrison B, Davis KP, Maddox JF. 1997. Ovine anonymous dinucleotide repeat polymorphism at the CSRD264, CSRD269, CSRD 270, CSRD 287, CSRD 2108, CSRD 2138 and CSRD 2164. Animal genetics, 28: 70 - 71.
Hulme DJ, Smith AJ, Silk JP, Redwin JM, Beh KJ. 1995. Polymorphic sheep microsatellites at the McM2, McM131, McM135, McM136, McM140, McM200, McM214, McM373, McM505, McM507 and McM512 loci. Animal Genetics, 26: 369 - 370.
Hulme, DJ, Silk JP, Redwin JM, Barendse W, Beh KJ. 1994. Ten polymorphic ovine microsatellites. Animal Genetics, 25: 434.
Israel C, Weller JI. 2000. Effect of misidentification on genetic gain and estimation of breeding value in dairy catlle populations. Journal of Dairy Science, 83: 181-187.
Kalinowski ST, Taper ML, Marshall TC. 2007. Revising IHow the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 16: 1099-1006.
Kelly L, Postiglioni A. 1997. Marcadores genéticos en equinos. I-Actualización de metodologías. Veterinaria, 33(135): 11-15.
Maddox JF, Davies KP, Crawford AM, Hulme DJ, Vaiman D, Cribiu EP, Freking BA, Beh KJ, Cockett NE, Kang N, Riffkin CD, Drinkwater R, Moore SS, Dodds KG, Lumsden JL, van Stijn TC, Phua SH, Adelson DL, Burkin HR, Broom JE, Buitkamp J, Cambridge L, Cushwa WT, Gerard E, Galloway SM, Harrison B, Hawken RJ, Hiendleder S, Henry HM, Medrano JF, Paterson KA, Schibler L, Stone RT, van Hest B. 2001. An enhanced linkage map of the sheep genome comprising more than 1000 loci. Genome Reserch, 11: 1275 -1289.
Raymond MF, Rousset F. 1995. GENEPOP (version 1.2): population genetics software for exact tests and ecumenicism.GENEPOP (versión 1.2): la genética de poblaciones de software para pruebas exactas y el ecumenismo. La herencia J, 86: 248 - 249.J. Heredity, 86:248-249.
Rousset F. 2008. Genepop’007: a complete reimplementation of the Genepop software for Windows and Linux. Molecular Ecology Resources, 8: 103-106.
Tilbrook AJ, Cameron AWN, Lindsay DR. 1987. The influence of ram mating preferences and social interaction between rams on the proportion of ewes mated at field joining. Applied Animal Behaviour Science, 18(2): 173-184.
Toldo SS, Fries R, Steffen P, Neibergs HL, Barendse W, Womack JE, Hetzel DJ, Stranzinger G. 1993. Physically mapped, cosmid-derived microsatellite markers as anchor loci on bovine chromosomes. Mammalian Genome, 4(12):720-727.
Tomasco I, Wlasiuk G, Lessa EP. 2002. Evaluation of polymorphism in ten microsatellite Loci in Uruguayan sheep flocks. Genetics and Molecular Biology, 25(1): 37-41.
Ungerfeld R, González-Pensado SP. 2008. Social rank affects reproductive development in male lambs. Animal Reproduction Science, 109: 161-171.
Weber JL, May PE. 1989. Abundant class of human DNA polymorphism which can be typed using the polymerase chain reaction. The American Journal of Human Genetics, 44(3): 388 - 396.