Services on Demand
Journal
Article
Related links
Share
Agrociencia (Uruguay)
Print version ISSN 1510-0839On-line version ISSN 2301-1548
Agrociencia Uruguay vol.17 no.1 Montevideo June 2013
Desarrollo de marcadores funcionales asociados a caracteres fenotípicos para identificación varietal en soja
Ibarra Mariela1, Castro Ariel2, Capdevielle Fabián3
1Instituto Nacional de Semillas (INASE). Camino Bertolotti s/n y ruta 8, km 29, Barros Blancos, Canelones, Uruguay. Correo electrónico: mibarra@inase.org.uy.
2Universidad de la República, Facultad de Agronomía, Estación Experimental Mario A. Cassinoni (EEMAC). Departamento de Producción Vegetal. Ruta 3 General Artigas km 363, 60000, Paysandú, Uruguay.
3Instituto Nacional de Investigación Agropecuaria. Unidad de Biotecnología. Estación Experimental INIA Las Brujas. Ruta 48 km 10, Rincón del Colorado, Canelones, Uruguay.
Recibido: 26/5/11 Aceptado: 21/1/13
Resumen
La organización de los sistemas agrícolas requiere la verificación de la identidad y pureza genética de los cultivares. El aumento en el número de cultivares de soja a ser evaluados y su estrecha base genética dificulta su identificación mediante evaluación de descriptores fenotípicos. La Unión Internacional para la Protección de las Obtenciones Vegetales (UPOV) ha reconocido la utilidad de marcadores moleculares asociados con caracteres fenotípicos descriptivos. Con el objetivo de desarrollar ese tipo de marcadores, se seleccionaron in silico seis SSR génicos o genómicos (Sat286, Satt229, GmPrx1, GMES1173, Satt571 y GmHi), más dos marcadores previamente reportados (GmF35H y SoyF3H). Todos fueron evaluados en 35 cultivares de soja. Los SSR seleccionados para peroxidasa en testa y color de hilo (GmPrx1 y GmHi) fueron monomórficos. El contenido de información polimórfica (PIC) promedio del conjunto de marcadores polimórficos fue 0,48, con una media de 3,12 alelos por locus. GmF35H discriminó las variedades de acuerdo al color de flor (blanca y violeta). El análisis discriminante determinó alto porcentaje de clasificación correcta para el carácter hábito de crecimiento (95,8%) y color de pubescencia (80,6%), utilizando Sat286 y SoyF3H respectivamente. Los valores de clasificación para color de vaina (74,2%) y tamaño de folíolo (73,5%) fueron intermedios, utilizando GMES1173 y Satt571 respectivamente. El marcador Satt229 no resultó discriminante para ciclo a floración (50%) y maduración (42,8%). Los marcadores moleculares seleccionados dentro o cercanos a secuencias de interés pueden ser integrados a un sistema de identificación genética de variedades de soja como complemento a los descriptores fenotípicos clásicos.
Palabras clave: GLYCINE MAX L., CRIBADO DE SSR, CARACTERIZACIÓN MOLECULAR
Summary
Development of Functional Markers Associated with Phenotypic Traits for Variety Identification in Soybean
The organization of agricultural systems requires the verification of the genetic identity and purity of cultivars. The increase in the number of soy varieties to be evaluated, and the narrow genetic base of soybean cultivars, make the identification using phenotypic descriptors very difficult. The International Union for the Protection of New Varieties of Plants (UPOV) has recognized the utility of molecular markers associated with descriptive phenotypic characteristics. With the goal of developing this kind of markers, six genic or genomic SSR were selected in silico (Sat286, Satt229, GmPrx1, GMES1173, Satt571 and GmHi), plus two previously reported markers (GmF35H and SoyF3H). All were evaluated in 35 soybean cultivars. The SSRs GmPrx1 and GmHi selected for seed coat peroxidase and hilum color respectively were monomorphic. The mean Polymorphism Information Content (PIC) value within the selected group of polymorphic markers was 0.48 with an average of 3.12 allele per locus. GmF35H discriminated the soybean varieties according to the flower color (white and purple). Discrimination tests showed a high percentage of accurate classification of growth habit (95.8%) and pubescence color (80.6%) with Sat286 and SoyF3H, respectively. The classification values for pod color (74.2%) and leaflet size (73.5%) were intermediate using GMES1173 and Satt571, respectively. The marker Satt229 was not discriminating for flowering time (50%) and maturity (42.8%). Molecular markers selected in or close to sequences of interest can be integrated into a genetic identification system as complementary markers to the classic phenotypic descriptors of soybean varieties.
Key words: GLYCINE MAX L., SSR MINING, MOLECULAR CHARACTERIZATION
Introducción
En los últimos años el cultivo de soja (Glycine max L.) ha tenido un gran crecimiento en Uruguay, con más de 863 mil hectáreas sembradas en el año 2009. Uruguay cuenta con 108 variedades de soja inscritas en el Registro de Propiedad de Cultivares, de las cuales 81 se encuentran a nivel comercial (INASE, 2010).
La Unión Internacional para la Protección de las Obtenciones Vegetales (UPOV), de la cual Uruguay es miembro (Uruguay. Poder Lesgislativo, 1994), constituye un sistema de propiedad intelectual aplicable a variedades vegetales. Los requisitos para la concesión de los derechos de propiedad incluyen la realización de ensayos parcelarios y plantas individuales a campo e invernáculo, con el objetivo de determinar la diferenciación, homogeneidad y estabilidad (DHE) de las nuevas variedades (UPOV, 2002a).
Las variedades de soja que se siembran en Uruguay provienen mayoritariamente de Argentina y Estados Unidos, y dada su estrecha base genética se dificulta la discriminación mediante evaluación fenotípica (Giancola et al., 2002). Esto, sumado al aumento constante en el volumen de muestras a ser testadas, hace atractiva la búsqueda de herramientas alternativas como las técnicas moleculares para la caracterización y discriminación varietal.
Numerosos sistemas de marcadores moleculares han sido evaluados como herramientas aplicables para la identificación de variedades en soja (Diwan y Cregan, 1997; Song et al., 1999; Priolli et al., 2002; Giancola et al., 2002). Este tipo de marcadores posee la ventaja de ser poco influenciado por el ambiente, está disponible en un número casi ilimitado y puede ser utilizado en etapas tempranas (semillas y plántulas) (Cooke, 1999).
Si bien UPOV no admite la identificación de una variedad solamente por medio de una comparación con marcadores moleculares neutros o «anónimos», se ha establecido la utilidad del uso de marcadores moleculares asociados a la expresión de caracteres fenotípicos (UPOV, 2002b).
Los marcadores de tipo microsatélite o SSR son marcadores moleculares altamente polimórficos, co-dominantes y altamente reproducibles. Han sido ampliamente utilizados para estudios de diversidad e integración de mapas genéticos en soja (Cregan et al., 1999; Narvel et al., 2000; Song et al., 2004). Generalmente se encuentran en zonas no codificantes del ADN por lo que pueden denominarse neutros, anónimos o genómicos, ya que no se asocian a ningún tipo de expresión fenotípica (Andersen y Lubberstedt, 2003; Gupta et al., 2003; Varshney et al., 2005a).
Variaciones en marcadores localizadas dentro de genes pueden implicar cambios fenotípicos por modificar la expresión o función de un gen al cual está asociado. En estos casos se han denominado marcadores funcionales (MF) (Andersen y Lubberstedt, 2003) y podrían ser utilizados para la identificación varietal. En soja se han mapeado un gran número de loci de efecto cuantitativo (QTLs por su sigla en inglés) asociados a caracteres de calidad, reproductivos, crecimiento y desarrollo. La localización de genes candidatos en el intervalo de QTLs que afectan fenotipos específicos constituye un recurso de desarrollo potencial de marcadores funcionales (Gupta y Rustgi, 2004; Varshney et al., 2005b). En este caso la probabilidad de éxito de encontrar genes relacionados con la característica depende de la resolución del intervalo del QTL. En general con intervalos menores a 3cM se podría comenzar a explorar secuencias (Borevitz y Chory, 2004), siendo más efectivo cuando la resolución es menor a 1cM (Ni et al., 2009).
Utilizando bases de datos de secuencias expresadas (ESTs) y datos de mapeo de QTLs, se podrían identificar genes relacionados a caracteres de interés, utilizando el escaneo de las secuencias de genes, ESTs y clones de ADNc para la búsqueda y cribado de microsatélites génicos in silico (Varshney et al., 2005c).
El objetivo del presente trabajo es determinar la asociación existente entre un conjunto de marcadores moleculares (génicos y genómicos) y caracteres fenotípicos de interés descriptivo, que permitan la discriminación de un grupo de 35 variedades de soja utilizadas en Uruguay. Para ello se utilizaron un conjunto de SSR y un marcador alelo específico, localizados dentro de QTLs de interés.
Materiales y métodos
Variedades y caracteres fenotípicos seleccionados
Se utilizaron 35 variedades de soja (Glycine max L.) pertenecientes a la colección de referencia del Instituto Nacional de Semillas (INASE), Uruguay.
Los caracteres seleccionados para búsqueda de marcadores asociados fueron seleccionados a partir de descriptores de UPOV e INASE (UPOV, 1998). Estos son: color de flor, color de pubescencia, color de vaina, hábito de crecimiento, peroxidasa en testa, color de hilo, forma de hoja, grupo de madurez y ciclo a floración. La información fenotípica de cada variedad proviene de datos de ensayos en INASE, durante 2007 y 2008.
Selección de marcadores
Se validaron experimentalmente dos marcadores funcionales: un marcador alelo específico (GmF35H) desarrollado por Zabala y Vodking (2007), determinante del color de flor, y un SSR génico (SoyF3H) reportado por Toda et al. (2005), asociado al color de pubescencia. Para los caracteres hábito de crecimiento, forma de hoja, color de vaina, color de hilo, peroxidasa en testa, ciclo a floración y maduración, los marcadores funcionales candidatos fueron desarrollados y seleccionados in silico.
El desarrollo de los marcadores candidatos se inició realizando una búsqueda e integración de referencias bibliográficas e información genómica para determinar QTLs y secuencias genómicas candidatas (genes y ESTs) seleccionadas del National Center for Biotechnology Information (NCBI:http://www.ncbi.nlm.nih.gov) y Soybase (http://www.soybase.org). A partir de fenotipos, se identificaron secuencias mediante búsquedas ontológicas en Gene Ontology Consortium (http://www.geneontology.org) y secuencias vinculadas a rutas metabólicas en Kyoto Encyclopedia of Genes and Genomes (KEGG: http://www.genome. jp/kegg). Se exploraron regiones de QTLs de soja en base a la información genómica de especies modelo (Arabidopsis thaliana) mediante BLAST en NCBI. Los genes seleccionados en la especie modelo se utilizaron para identificar ortólogos en soja Soybase (http://www.soybase.org) y Phytosome (http://www.Phytozome.net). En la selección se priorizaron SSR génicos, localizados dentro de genes cuyo producto hipotético se relacionó a procesos metabólicos influyentes en la determinación del carácter. En segundo lugar se consideraron los SSR genómicos localizados dentro del intervalo definido para QTLs vinculados con el carácter seleccionado.
Para detectar los SSR en las secuencias seleccionadas se utilizó el programa CUGISSR (Clemson University Genome Institute, USA). Los SSR seleccionados corresponden a dinucleótidos, trinucleótidos, tetranucleótidos con más de 11, 6 y 4 repeticiones respectivamente.
Los cebadores fueron sintetizados por el servicio de IDT (Integrated DNA Technologies, Inc. USA). Se seleccionaron siete marcadores SSR y un marcador alelo específico, para su evaluación experimental en las 35 muestras de soja (Cuadro 1).
Extracción de ADN vegetal
El ADN fue extraído de acuerdo al método CTAB (Bromuro de cetiltrimetilamonio) (Murray y Thompson, 1980) de una muestra de tejido foliar fresco de 0,1 g obtenido de hojas jóvenes de 15 plantas de cada variedad. La concentración y calidad de ADN obtenido se determinó en comparación con patrones de bandas de concentración conocida mediante electroforesis en gel de agarosa al 1% y tinción con bromuro de etidio.
Amplificación y detección de polimorfismos
Para detección de los SSR se utilizaron cebadores con marcación fluorescente M13 para la visualización de los fragmentos mediante la utilización de fluorocromos incorporado en los productos amplificados (Schuelke, 2000). La mezcla para reacción de PCR para un volumen final de 11 µL consistió en: 7,58 µL de agua Milli-Q, 1,5 µL Buffer (10X), 0,33 µL MgCl2 (50 mM), 0,22 µL dNTPs (10mM), 0,11 µL primer M13 (10 µM), 0,11 µL primer reverse (10 µM), 0,05 µL primer forward+M13 (10 µM), 0,1 µL Taq polymerase (5U) (Invitrogen, Life Technologies Corporation, Carlsbad, CA, USA) y 1 µL de ADN. Las condiciones de PCR utilizadas fueron: 3 min a 94 °C, seguido por 35 ciclos de: 94 °C por 30 s, 54 °C por 1 min, y 72 °C por 2 min. Las temperaturas de anidamiento de 48 a 55 °C dependiendo de los cebadores utilizados. Al protocolo anteriormente descripto se le incorporó un protocolo de ciclado de acuerdo a Schuelke (2000) para favorecer la amplificación del cebador M13. Las condiciones de PCR adicionales consistieron en ocho ciclos de: 94 °C por 30 s, 53 °C por 45 s, y 72 °C por 45 s y un período final de extensión de 2 min a 72 °C. Las amplificaciones fueron realizadas en un termociclador MultiGene con gradiente térmico (Labnet International Inc., NJ, USA). La calidad de amplificación de los productos de PCR se verificó mediante electroforesis con 5 µl del producto PCR en geles de agarosa al 2 % y visualizados en solución con bromuro de etidio. El genotipado de los productos de amplificación fue realizado en el servicio de análisis de DNA de la Universidad de Yale, USA. El marcador alelo específico GmF35H, fue amplificado según las condiciones descritas por Zabala y Vodking (2007) y visualizado en gel de agarosa al 3%.
Análisis de datos
El análisis de la presencia y tamaño de los fragmentos obtenidos se realizó utilizando el software Peak Scanner v1.0 (Applied Biosystems, Life Technologies Corporation, Carlsbad, CA, USA). El contenido de información polimórfica (PIC) de los marcadores se analizó con el paquete estadístico Cervus 3.0.3 (Field Genetics Ltd. www.fieldgenetics.com) (Kalinowski et al. 2007). Se utilizó la fórmula establecida por Botstein et al. (1980) y Hearne et al. (1992). Este índice se define como:
PIC = 1- Σpi2, siendo pi la frecuencia del alelo i-ésimo.
Para integrar la información de marcadores génicos y genómicos asociados a caracteres fenotípicos se utilizaron análisis discriminantes. Con los datos de marcadores se elaboró una matriz de variables discretas de 0 y 1, para presencia y ausencia de cada alelo por marcador. Se realizó un análisis de clasificación utilizando el algoritmo «vecino más cercano» (IBK) implementado en WEKA (versión 3.6.3. Waikato Environment for Knowledge Analysis, University of Waikato, Hamilton, NZ, http://www.cs.waikato.ac.nz/~ml/Weka) (Witten y Frank, 2005). Los análisis de clasificación se realizaron entre las clases fenotípicas presentes para cada carácter, utilizando la información molecular obtenida. Los resultados se expresan como porcentaje de clasificación correcta para cada variedad de acuerdo a su correspondiente clase fenotípica.
Para medir la capacidad de acierto de la clasificación se utilizó el coeficiente estadístico Kappa (Cohen, 1960).
Kappa = Pr(a) – Pr(e)
1 – Pr(e)
donde: Pr(a) es la proporción de aciertos observados y Pr(e) es la probabilidad esperada de aciertos por azar. Kappa permite estimar si la clasificación obtenida puede ser producto del azar (k ≤ 0) así como el grado de robustez del clasificador utilizado (mayor cuanto más cercano a 1).
Resultados y discusión
Selección y desarrollo de marcadores asociados a caracteres fenotípicos
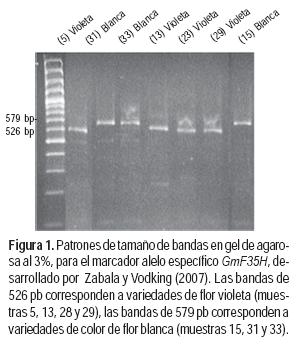
En soja el color de flor (blanca o violeta) está determinado por el gen W1 en el cromosoma 13. Se seleccionó el marcador GmF35H previamente reportado por Zabala y Vodking (2007) amplificando fragmentos de ADN de 526-bp y 579-bp en 18 cultivares e isolíneas W1 (púrpuras) o w1 (blancas) respectivamente.
El color de pubescencia gris (PG) o tostado (PT) es determinado por un gen (T), codificando la proteína flavonoide 3´hidroxilasa (Flavonoid 3´ hidroxilase) (Toda et al., 2002). Dentro de la secuencia del gen SoyF3´H se determinó la presencia de un microsatélite dinucleótido polimórfico en variedades de PG y PT (Toda et al., 2005).
El color de vaina es un marcador morfológico (L2) mapeado en la posición cero del cromosoma 3 (http://www.soybase.org). Para la selección de secuencias candidatas se realizó una búsqueda de genes y ESTs por los términos de anotación que incluyó Flavonoides (antocianinas y proantocianidinas) en KEGG (Winkel, 2001; Pourcel et al., 2006). De los 16 SSR-EST obtenidos, dos se ubicaron dentro de la región cercana a L2. Se seleccionó el marcador GMES1173 (AAC17), correspondiente al gen Glyma03g00410 con anotación hipotética tiorredoxina (Thioredoxin-like proteín) a una distancia aproximada de 2 cM de L2 (Cuadro 1).
El principal QTL para hábito de crecimiento (DT1) en soja explica el 72,5% de la variación (Cregan et al., 1999). Para realizar una aproximación a los genes candidatos de esta característica en soja, se realizó una búsqueda basada en ontologías vegetativo-reproductivas para la especie modelo Arabidopsis thaliana (TAIR). Mediante el algoritmo BLAST (NCBI) se determinó la homología del gen Centroradialis/Terminal Flower 1-like (GmTFL1) de Arabidopsis con el gen de soja Glyma19g37890.1 con anotación similar (Cuadro 1). Estudios recientes confirman a este gen en el locus Dt1 como determinante del carácter hábito de crecimiento en soja (Liu et al., 2010; Tian et al., 2010). En la exploración de la secuencia ampliada hacia regiones cercanas al gen se determinó el Sat286 como el marcador más próximo al gen (910 pb) (http://www.soybase.org), siendo seleccionado para evaluación experimental.
Como forma de aproximación para la selección de marcadores funcionales asociados a forma y tamaño de hoja en soja, la búsqueda por ontología de palabras claves (http://www.geneontology.org) obtuvo como resultado cinco productos génicos de Arabidopsis. Dichas secuencias fueron comparadas en bases de datos de ESTs mediante el algoritmo BLAST en NCBI. Se obtuvieron 168 secuencias ESTs con anotación hipotética, de los cuales se obtuvieron 21 SSR-ESTs y tres SSR génicos para los términos de anotación. Para evaluación experimental se seleccionó un SSR dentro del gen Glyma20g01760.1 del cromosoma 20, con anotación funcional similar a PIN1 proteína de transporte de auxina (auxin transport proteín). Este microsatélite corresponde al Satt 571 (GmPin1, en este trabajo) y se encuentra en la zona de dos QTLs mapeados para forma y largo de folíolo (Lflgth7-9 y Lflt shape 7-7) (http://www.soybase.or).
En soja se han determinado al menos siete genes incluído E3 que determinan el momento de floración y maduración (Buzell y Voldeng, 1980; McBlain y Bernard, 1987; Cober y Voldeng, 2001). Liu et al. (2008) reportaron la asociación entre el fitocromo A y la sensibilidad al fotoperíodo. Adicionalmente, Molnar et al. (2003) determinaron el SSR Satt229 como el marcador más próximo al locus E3. El SSR Satt229 mapea en el gen Glyma19g41210 con anotación histidina quinasa (histidine kinase) (correspondiendo al gen GmPhyA3 del fitocromo A de Glycine max). En este estudio, Satt229 se utilizó como candidato para su análisis funcional en variedades registradas en Uruguay.
La reacción de la peroxidasa en la testa de semillas de soja produce la presencia o ausencia de coloración útil para discriminar variedades, se seleccionó el SSR GmPrx1 dentro de la secuencia AF014502 (http://www.ncbi.nlm. nih.gov) correspondiente al gen Ep, Glyma09g02590 precursor de la peroxidasa en testa.
Para la selección de SSR candidatos para color de hilo se determinó la ubicación del locus I en el cromosoma 8 de soja. En dicha región se ubican dos genes glyma08g11520 y glyma08g11530 (http://soybase.org/gbrowse/cgi-bin/gbrowse), ambas corresponden a enzimas chalcone sinteasa involucrada en la síntesis de flavonoides y en cuya región se ubica el gen que afecta el color de hilio y el color de testa (Palmer et al., 2004). Se seleccionó un SSR AT (26) GmHi ubicado a 260pb aproximadamente del gen Gyma08g11530, locus FJ770471 (http://www.ncbi.nlm.nih.gov/nucleotide/FJ770471.1).
Evaluación de marcadores
Para los microsatélites seleccionados no se obtuvo resultados de genotipado en la totalidad de las 35 variedades (Cuadro 2), la falta de resultados de amplificación en algunas muestras podría explicarse por baja calidad del ADN extraído para secuenciar o a la presencia de alelos nulos.
De los ocho marcadores analizados, seis amplificaron productos polimórficos con un total de 23 alelos (Cuadro 2). El marcador génico GmPrx1 y el genómico GmHi, seleccionados para peroxidasa en testa y color de hilo respectivamente, fueron monomórficos para las variedades analizadas. El rango de alelos en los SSR fue de tres a seis, con una media de 3,13 alelos por locus. El PIC promedio del conjunto de marcadores fue de 0,48, variando de 0,37 para el marcador alelo específico F35H, a 0,64 para el SSR genómico Satt286 (Cuadro 2).
El bajo nivel de polimorfismo detectado puede en parte ser explicado por la estrecha base genética de las variedades de soja cultivadas. Se estima que un grupo reducido de 17 variedades ancestrales norteamericanas (accesiones de origen asiático) contribuyen con el 86% de los genes presentes en el pool de genes de cultivares de Norte America (Gizlice et al., 1994). En variedades argentinas, Giancola et al. (2002) determinaron índices de diversidad genética medios (0,6) utilizando SSR genómicos en 100 variedades de soja. Otros trabajos presentan valores diversidad alélica entre 0,6 y 0,7 en promedio en variedades elite (Song et al., 2004, 1999; Narvel et al., 2000; Arnao et al., 2010). Los mayores niveles de polimorfismo > 0,7 se reportan en trabajos donde se utilizan SSR genómicos o neutros altamente polimórficos en variedades provenientes de centros de origen o silvestres (Hudcovicová y Kraic, 2003; Hua et al., 2005; Yoon et al. 2009).
La mayoría de los marcadores utilizados en este trabajo son derivados de genes o secuencias expresadas (SSR génicos), que presentan menores niveles de polimorfismo que SSR genómicos o neutros (Song et al., 2004). Esto puede deberse a que variaciones registradas dentro de las secuencias reguladoras, UTR y marcos de lectura (ORF) pueden tener efectos deletéreos (Gupta et al., 2003; Song et al., 2004). Por otra parte, los marcadores génicos por su importancia funcional pueden estar sujetos a selección en los procesos de mejoramiento genético por lo cual es esperable una menor variabilidad. Esta menor variabilidad se observa claramente en trabajos que evalúan ambos tipos de SSR (génicos y genómicos) en los mismos cultivares. Por ejemplo en soja, Mulato et al. (2010) evaluaron 10 marcadores SSR-ESTs obteniendo un PIC promedio de 0,45 y con 20 marcadores SSR genómicos determinó un PIC de 0,714 en 79 accesiones de diferentes orígenes. En el mismo sentido, Fu et al. (2007) determinaron una media de PIC de 0,69 con 23 SSR genómicos y una media de PIC de 0,54 con 14 SSR-ESTs en 82 variedades de soja canadienses.
Análisis de asociación
El marcador alelo específico GmF35H desarrollado por Zabala y Vodking (2007) permitió discriminar las 35 variedades testadas de acuerdo a su color de flor. La Figura 1 muestra como ejemplo el análisis de siete de las 35 variedades, de las cuales cuatro presentan flores violetas y tres flores blancas. Las variedades con flor blanca presentaron una banda correspondiente a productos de amplificación de 579 pb, mientras que las de flor violeta presentaron una banda aproximada de 526 pb, siendo los resultados similares a los a los autores.
Para color de pubescencia, utilizando el marcador SSR génico F3H (Toda et al., 2005), se identificaron cinco alelos en 31 variedades amplificadas (Cuadro 2).El análisis discriminante obtuvo un 80,65% de clasificación correcta para las clases de color de pubescencia tostado (PT) y gris (PG), con un índice de Kappa de 0,6157 (Cuadro 3). Once de las doce variedades de PT presentaron el alelo de 192 pb y la variedad restante presentó un alelo único de 202 pb. En su mayoría, 11 de 19 variedades de PG presentaron el alelo de 211 pb, mientras que las restantes presentaron alelos de 192, 194 y 204 pb. Los rangos de tamaño de alelos son similares a los obtenidos por Toda et al. (2005), donde utilizando dos pares de líneas NILs y seis cultivares de diferentes orígenes determinó rangos desde 171pb a 185pb para variedades de pubescencia tostada y de 159pb a 205 pb en variedades y líneas con pubescencia gris.
Para el marcador GMES1173, seleccionado para color de vaina, presentó locus heterocigoto con el alelo de 220 bp en todas las muestras analizadas. El porcentaje de clasificación correcta fue de 74,2% (Kappa, 0.4918) entre los tres alelos del marcador y las clases de color de vaina clara y oscura presentes en las variedades testadas (Cuadro 3). Doce de las catorce muestras de vaina clara presentaron el alelo 230bp, mientras que once de las diecisiete de vaina oscura presentaron el alelo 243bp.
Para el SSR Sat286, se determinaron seis alelos de diferentes tamaños -entre 164 y 180 pb- en 24 variedades genotipadas. En el análisis de asociación con este marcador, se obtuvo un 95,8% (23 de 24 variedades) de clasificación correcta en sus clases fenotípicas (Kappa 0,8947) (Cuadro 3). La mayoría de las variedades de tipo indeterminado (13 de 18) presentaron el alelo de 178pby en menor proporción los alelos 176pb y 180pb. Las seis variedades de tipo determinado clasificaron correctamente, presentando los alelos 164, 166 y 168pb. La muestra clasificada en la clase incorrecta corresponde a una variedad tipo indeterminado que presentó el alelo de 166 pb presente en la mayoría de las variedades determinadas. Este alelo también está presente en una variedad de fenotipo indeterminado con el locus heterocigoto con 166/178pb, siendo clasificada correctamente en su categoría.
En este estudio no se determinó asociación entre el SSR génico Satt229 con ciclo a floración ni con ciclo a madurez. Dado el ligamiento existente entre E3 y Dt1, no se encontró una asociación significativa entre alelos de Sat286 y las clases fenotípicas vinculadas con floración y maduración (datos no mostrados).
El análisis de asociación del marcador Satt571 (GmPin1) entre las cuatro clases fenotípicas del carácter FF presentó un bajo porcentaje de clasificación 55,9%, con un índice Kappa negativo de -0,0429. Sin embargo, este marcador presentó un 73,5% de asociación con el TF, para las clases pequeño, mediano y grande, con un Kappa de 0,3077 (Cuadro 3).
Dado que uno de los principales QTLs para forma y tamaño de foliolo se encuentra en el cromosoma 19, cerca de Dt1 y E3 (Yamanaka et al., 2001), se realizaron análisis de asociación de las clases fenotípicas TF y FF con los marcadores Sat286 y Satt229 mapeados en dicha región cromosómica (http://www.soybase.org). Se determinó un alto porcentaje de clasificación correcta (83,33%, Kappa 0,53) entre el marcador Sat286 y tamaño de folíolo. En las 24 variedades genotipadas, los tres genotipos de folíolo pequeño y un genotipo de folíolo grande fueron incorrectamente clasificadas en la clase media. Se determinó bajo porcentaje de asociación entre el marcador Satt229 y las características TF y FF (Cuadro 3).
Finalmente se realizó el análisis de la capacidad discriminante para el set de seis marcadores polimórficos determinados en este estudio. Dicho set discriminó el 65,7% de los cultivares, correspondiendo a 23 de las 35 variedades de soja en este estudio, con un Kappa de 0,6471 (Cuadro 3).
Este set de marcadores por sí solo no resulta capaz de discriminar las 35 variedades de soja evaluadas, lo que es probable que se deba al bajo nivel de polimorfismo detectado, al número reducido de marcadores y cultivares evaluados y la probabilidad de existencia de alelos nulos (no fue verificada en este trabajo), por la falta de amplificación de algunos marcadores, disminuyendo el volumen de información analizada. En este trabajo se determinaron algunas asociaciones de interés. El marcador GmF35H discriminó las 35 variedades de soja de acuerdo al color de flor. Utilizando análisis discriminantes se determinó un alto porcentaje de clasificación correcta para el carácter hábito de crecimiento (95,8%) y color de pubescencia (80,6%), con los SSRs Sat286 y SoyF3H respectivamente. Los valores de clasificación correcta obtenidos para color de vaina (74,2%) y tamaño de folíolo (73,5%) fueron intermedios, utilizando los SSRs GMES1173 y Satt571 (GmPin1) respectivamente. Los resultados son promisorios. No obstante, las mismas no son suficientes para establecer un set mínimo de marcadores funcionales capaces de discriminar la totalidad de los genotipos evaluados.
Con los datos obtenidos no alcanza para generar un sistema basado en marcadores moleculares que sustituya a los marcadores fenotípicos. La mayoría de los marcadores evaluados no son 100% efectivos en determinar diferencias fenotípicas. La complejidad de los mecanismos genéticos involucrados en algunos de los caracteres evaluados disminuye la posibilidad de encontrar asociaciones de interés utilizando pocos marcadores candidatos. Además hay que considerar que en algunos casos las clases fenotípicas están formadas por variables de tipo cuantitativas (ejemplo días a floración y maduración) y que el criterio de definición de clase es muchas veces arbitrario, por lo cual es esperable variación alélica dentro de la clase fenotípica.
Si bien los marcadores moleculares no sustituyen a los marcadores morfológicos, contar con marcadores asociados a caracteres fenotípicos descriptivos posee varias ventajas y aplicaciones como herramientas complementarias para el registro y contralor de variedades. Por ejemplo son de utilidad para evaluar caracteres morfológicos difíciles de evaluar fenotípicamente como algunos tipos de resistencias a estrés biótico o abiótico, caracteres de calidad, etc. En otros casos, los caracteres morfológicos son fácilmente evaluables pero se manifiestan tardíamente en el ciclo de las plantas y los ensayos requieren de un mantenimiento continuo, estando expuestos a condiciones climáticas adversas que pueden comprometer el óptimo desarrollo de las plantas.
Disponer de herramientas moleculares facilita la identificación temprana y preselección de muestras a ser sembradas en ensayos de identificación y discriminación varietal. Además son de utilidad como criterio de selección previo de variedades a ser sembradas en ensayos de verificación varietal en muestras de identidad desconocida, permitiendo acotar el número de muestras referencia contra las cuales deben ser testadas a campo, con la consiguiente disminución de tiempo y costos.
Conclusiones
Este trabajo constituye una aproximación al desarrollo y utilización potencial de marcadores funcionales asociados a características de interés descriptivo. Si bien el número de cultivares y marcadores analizados es limitado, los resultados obtenidos para algunos de los marcadores funcionales son prometedores y merecen el estudio en el futuro en un número más amplio de accesiones. Los resultados validaron, en un grupo de variedades comerciales de soja sembradas en Uruguay, las asociaciones entre el marcador F35H y el color de flor, y el marcador F3H y el color de pubescencia. Se identificó un marcador SSR genómico altamente discriminante para el carácter hábito de crecimiento (Sat286) y otros dos SSR con niveles intermedios de discriminación para caracteres morfológicos: Satt571 (GmPin1) con forma de hoja y GMES1173 con color de vaina. Estos marcadores fueron seleccionados dentro o cercanos a la secuencia de genes que expresan proteínas relacionadas con las rutas metabólicas que afectan la expresión del carácter. Este grupo de cinco marcadores (F35H, F3H, Sat286, Satt571 (GmPin1) y GMES1173) podría ser incorporado como descriptores varietales asociados a caracteres fenotípicos en base al análisis que se realice en el marco de UPOV.
El conjunto de marcadores seleccionados podrían integrarse a un sistema de identificación genética de variedades de soja como complemento a los descriptores fenotípicos clásicos.
Bibliografía
Andersen JR, Lubberstedt T. 2003. Functional markers in plants. Trends in Plant Science, 8: 554 - 560.
Arnao E, Perdomo R, Graterol E. 2010. Diversidad genética en cultivares de soya utilizando marcadores microsatellites en Venezuela. Interciencia, 35 (7): 534 - 538.
Borevitz JO, Chory J. 2004. Genomics tools for QTL analysis and gene discovery. Current Opinion in Plant Biology, 7: 132 -136.
Botstein D, White R, Skolnick M, Davis R. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 32: 314 -331.
Buzzell RI, Voldeng HD. 1980. Inheritance of insensitivity to long daylength. Soybean Genetics Newsletter, 7: 26 - 29.
Cober ER, Voldeng HD. 2001. A new soybean maturity and photoperiod-sensitivity locus linked to E1 and T. Crop Science, 41: 698 -701.
Cohen J. 1960. A coefficient of agreement for nominal scales. Educational and Psychological Measurement, 20: 37-46.
Cooke RJ. 1999. Modern methods for the cultivar identification and the transgenic plant challenge. Seed Science and Technology, 27: 669-680.
Cregan PB, Jarvik T, Bush A, Shoemaker R, Lark K, Kahler A, Kaya N, VanToai T, Lohnes D, Chung J, Specht. J. 1999. An integrated genetic linkage map of the soybean genome. Crop Science, 39: 1464 -1490.
Diwan N, Cregan PB. 1997. Automated sizing of fluorescent labeled simple sequence repeat (SSR) markers to assay genetic variation in soybean. Theoretical and Applied Genetics, 95: 723-733.
Fu Y-B, Peterson G, Morrison M. 2007. Genetic Diversity of Canadian Soybean Cultivars and Exotic Germplasm Revealed by Simple Sequence Repeat Markers. Crop Science, 47: 1947-1954.
Giancola S, Poltri S, Lacaze P, Hopp H. 2002. Feasibility of integration of molecular markers and morphological descriptors in a real case study of a plant variety protection system for soybean. Euphytica, 127: 95-113.
Gizlice Z, Carter T, Burton J. 1994. Genetic base for North American public soybean cultivars released between 1947 and 1988. Crop Science, 34: 1143 - 1151.
Gupta PK, Rustgi S. 2004. Molecular markers derived from expressed/transcribed portion of the genome in higher plants. Functional & Integrative Genomics, 4: 139 -162.
Gupta PK, Rustgi S, Sharma S, Singh R, Kumar N, Balyan H. 2003. Transferable EST-SSR markers for the study of polymorphism and genetic diversity in bread wheat. Molecular Genetics and Genomics, 270: 315 - 323.
Hearne C, Ghosh S, Todd J. 1992. Microsatellites for linkage analysis of genetic traits. Trends in Ecology and Evolution, 8: 288 - 294.
Hua X, Rongxia W, Ruzhen C, Lijuan Q. 2005. Genetic diversity of Chinese summer soybean germplasm revealed by SSR markers. Chinese Science Bulletin, 50(6): 526-535.
Hudcovicová M, Kraic J. 2003. Utilisation of SSRs for Characterisation of the Soybean (Glycine max (L.) Merr.) Genetic Resources. Czech Journal of Genetics and Plant Breeding, 39(4): 120-126.
INASE. 2010. Registro Nacional de Cultivares y Registro de Propiedad de Cultivares. [En línea]. Consultado setiembre 2010. Disponible en: http://www.inase.org.uy/especiesCultivares/Default.aspx.
Kalinowski S, Taper M, Marshall T. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 16: 1099 - 1106.
Liu B, Watanabe S, Uchiyama T, Kong F, Kanazawa A, Xia Z, Nagamatsu A, Arai M, Yamada T, Kitamura K, Masuta C, Harada K, Abe J. 2010. The Soybean Stem Growth Habit Gene Dt1 Is an Ortholog of Arabidopsis TERMINAL FLOWER1. Plant Physiology, 153: 198 -210.
Liu B, Kanazawa A, Matsumura H, Takahashi R, Harada K, Abe J. 2008. Genetic Redundancy in Soybean Photoresponses Associated With Duplication of the Phytochrome A Gene. Genetics, 180: 995 – 1007.
McBlain B, Bernard RL. 1987. A new gene affecting the time of flowering and maturity in soybean. Journal of Heredity, 78: 160 -162.
Molnar S, Rai S, Charette M, Cober E. 2003. Simple sequence repeat (SSR) markers linked to E1, E3, E4, and E7 maturity genes in soybean Genome, 46: 1024-1036.
Mulato BM, Möller M, Zucchi MI, Quecini V, Baldin J. 2010. Diversidade genética em germoplasma de soja identificada por marcadores SSR e EST-SSR. Pesquisa Agropecuária Brasileira, 45: 276 - 283.
Murray M, Thompson W. 1980. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Research, 8: 4321 - 4326.
Narvel J, Fehr W, Chu W, Grant D, Shoemaker R. 2000. Simple Sequence Repeat Diversity among Soybean Plant Introductions and Elite Genotypes. Crop Science, 40: 1452 -1458.
Ni J, Pujar A, Youens-Clark K, Yap I, Jaiswal P, Tecle I, Tung C-W, Ren L, Spooner W, Wei X, Avraham S, Ware D, Stein L, McCouch S. 2009. Gramene QTL database: development, content and applications. The Journal of Biological Databases and Curation, bap005.
Palmer RG, Pfeiffer TW, Buss GR, Kilen TC. 2004. Qualitative genetics. En: Boerma HR, Specht JE. [Eds.] Soybeans: Improvement, production, and uses. 3rd. ed. Madison : ASA, CSSA, SSSA. (Agronomy Monograph ; 16). pp. 137 - 233.
Priolli R, Mendes-Junior C, Arantes N, Contel E. 2002. Characterization of Brazilian soybean cultivars using microsatellite markers. Genetics and Molecular Biology, 25: 185 -193.
Pourcel L, Routaboul J, Cheynier V, Lepiniec L, Debeaujon I. 2006. Flavonoid oxidation in plants: from biochemical properties to physiological functions. Trends in Plant Science, 12: 1360 -1385.
Schuelke M. 2000. An economic method for the fluorescent labeling of PCR fragments. Nature Biotechnology, 18: 233 - 234.
Song Q, Marek L, Shoemaker R, Lark K, Concibido V, Delannay X, Specht J, Cregan P. 2004. A new integrated genetic linkage map of the soybean. Theoretical and Applied. Genetics, 109: 122 -128.
Song Q, Quigley C, Nelson R, Carter T, Boerma H, Strachan J, Cregan P. 1999. A selected set of trinucleotide simple sequence repeat markers for soybean cultivar identification. Plant Varieties and Seeds, 12: 207- 220.
Tian Z, Wang X, Lee R, Li Y, Spechtd J, Nelson R, McClean P, Qiu L, Ma J. 2010. Artificial selection for determinate growth habit in soybean. Proceedings of the National Academy of Sciences, 107(19): 8563-8568.
Toda K, Akasaka M, Dubouzet E, Kawasaki S, Takahashi R. 2005. Structure of Flavonoid 3´-Hydroxylase gene for pubescence color in soybean. Crop Science, 45: 2212 -2217.
Toda K, Yang D, Yamanaka N, Watanabe S, Harada S, Takahashi R. 2002. A single-base deletion in soybean flavonoid 3´-hydroxylase gene is associated with gray pubescence color. Plant Molecular Biology, 50: 187-196.
UPOV. 2002a. General introduction to the examination of distinctness, uniformity and stability and the development of harmonized descriptions of new varieties of plants [En línea]. Génova : UPOV. 26p. Consultado 19 marzo 2013. Disponible en: http://www.upov.int/en/publications/tg-rom/tg001/tg_1_3.pdf.
UPOV. 2002b. Ad hoc subgroup of technical and legal experts on biochemical and molecular techniques. Génova : UPOV. 5p.
UPOV. 1998. Guidelines for the conduct of tests for distinctness, homogeneity and stability (Sybean). Génova : UPOV. 39p.
Uruguay. Poder Legislativo. 1994. Ley Nº 16.580. Protección de las Obtenciones Vegetales. 21 setiembre de 1994.
Varshney RK, Graner A, Sorrells ME. 2005a. Genic microsatellite markers in plants: features and applications. Trends in Biotechnology, 23: 48-55.
Varshney RK, Graner A, Sorrells ME. 2005b. Genomics-assisted breeding for crop improvement. Trends in Plant Science, 10: 621- 630.
Varshney RK, Sigmund R, Borner A, Korzun V, Stein N, Sorrells ME, Langridge P, Graner A. 2005c. Interspecific transferability and comparative mapping of barley EST-SSR markers in wheat, rye and rice. Plant Science, 168: 195-202.
Winkel B. 2001. Flavonoid biosynthesis. A colorful model for genetics, biochemistry, cell Biology, and biotechnology. Plant Physiology, 126: 485-493.
Witten IH, Frank E. 2005. Data mining: practical machine learning tools and techniques. 2nd.ed. San Francisco: Morgan-Kufmann. 525p.
Yamanaka N, Ninomiya S, Hoshi M, Tsubokura Y, Yano M, Nagamura Y, Sasaki T, Harada K. 2001. An informative linkage map of soybean reveals QTLs for flowering time, leaflet morphology and regions of segregation distortion. DNA Research, 8: 61 – 72.
Yoon MS, Lee L, Kim CI, Kang JH, Cho EG, Baek HJ. 2009. DNA profiling and genetic diversity of Korean soybean (Glycine max (L.) Merrill) landraces by SSR markers. Euphytica, 165: 69 - 77.
Zabala G, Vodkin L. 2007. A rearrangement resulting in small tandem repeats in the F32 52 H genes of white flower genotypes is associated with the soybean W1 locus. Plant Genome, 2: 113 -124.