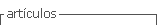Servicios Personalizados
Revista
Articulo
Links relacionados
Compartir
Agrociencia (Uruguay)
versión impresa ISSN 1510-0839versión On-line ISSN 2301-1548
Agrociencia Uruguay vol.16 no.1 Montevideo jun. 2012
Técnicas biológicas, serológicas y moleculares para la detección asintomática de Xanthomonas axonopodis pv. citri
Peyrou Mercedes1, Rigamonti Natalia2, Del Campo Raquel1, Russi Paola1, Larrechart Leticia3, Pérez Faggiani Elena3, Mara Héctor1
1Instituto de Investigaciones Biológicas Clemente Estable. Departamento de Biología Molecular. Avda. Italia 3318. C.P. 11600 Montevideo.
2 Instituto Nacional de Investigación Agropecuaria. Estación Experimental Wilson Ferreira Aldunate, Ruta 48 km 10 Las Brujas, Canelones.
3Instituto Nacional de Investigación Agropecuaria. Estación Experimental Salto Grande, Camino al Terrible s/n, Salto Grande, Salto. Correo electrónico: elenaperez@sg.inia.org.uy
Recibido: 21/6/11 Aceptado: 15/12/11
Resumen
Xanthomonas axonopodis pv. citri (Xac) produce cancro cítrico en todas las especies cítricas comerciales. La bacteria puede diseminarse mediante material vegetal de propagación contaminado y asintomático. Con el objetivo de optimizar técnicas de detección de bacterias para el análisis de rutina de material vegetal cítrico asintomático, se compararon las técnicas ELISA, Inmunoflorescencia, PCR, qRT_PCR e inoculación en plantas indicadoras (bioensayo). Las pruebas se realizaron a partir de diluciones al décimo entre 108 ufc.mL-1 y 102 ufc.mL-1 de un cultivo puro de cepa 49b. El nivel de detección obtenido fue de 1,8 x 102 ufc. mL-1 utilizando inmunoflorescencia, 1,8x104 ufc.mL-1 con ELISA indirecto, 1,8x103 ufc. mL-1 mediante la técnica de PCR, 10 ufc. mL-1 a través de qRT_PCR, y 230 ufc.mL -1 en plantas inoculadas de naranjo amargo. En base a estos resultados y teniendo en cuenta que para el análisis masivo de muestras es importante además de la sensibilidad, el costo y la practicidad, las técnicas de PCR e inoculación en plantas hospedero fueron las que reunieron las mejores características para ser evaluadas en material vegetal asintomático.
Palabras clave: CANCRO CÍTRICO, DIAGNÓSTICO, ELISA, PCR, BIOENSAYO
Summary
Biological, Serological and Molecular Techniques to Xanthomonas Axonopodis pv. Citri Asymptomatic Detection
Xanthomonas axonopodis pv. citri (Xac) produces citrus canker disease in all citrus commercial species. The bacteria can be disseminated through vegetative propagation material in asymptomatic form. To optimize bacteria detection techniques applicable to asymptomatic citrus plant tissue routine analysis, ELISA, Immunofluorescence, PCR, qRT_PCR and host plant inoculation (bioassay) diagnostic techniques were compared. Tests were made from decimal dilutions between 108 ufc.mL-1 and 102 ufc.mL-1 using a pure culture of 49b strain.The detection level obtained was 1.8 x 102 ufc.mL-1 using Inmunofluorescence; 1.8x104 ufc.mL-1 with indirect ELISA, 1.8 x 103 ufc.mL-1 by means of PCR; 10 ufc.mL-1 through of qRT_PCR and 230 ufc.mL-1 in sour orange inoculated plants. The experiment was repeated at least three times for each technique. Considering this result, and taking into account that sensitivity, practicity and cost, are important when a great number of plants need to be tested, the PCR and inoculation in host plants were those that met the best characteristics to be evaluated in asymptomatic plant material.
Key words: CITRUS CANKER, DIAGNOSTIC, ELISA, PCR, BIOASSAY
Introducción
El cancro cítrico producido por Xanthomonas axonopodis pv. citri (Xac) es una enfermedad que afecta a todas las especies cítricas comerciales, produciendo pérdidas en rendimiento y calidad de la fruta (Graham et al., 2004). Sin embargo el problema principal son las restricciones cuarentenarias impuestas por los países importadores a los productos provenientes de zonas donde la enfermedad está presente. Para permitir su ingreso, deben cumplirse condiciones (López, 1996), tales como la ausencia absoluta de fruta con síntomas de cancro cítrico, ya que la presencia de una fruta con lesiones determina el rechazo de la partida. Esta condición requiere estrategias de manejo muy exigentes y costosas y vuelve imprescindible evitar el ingreso de la enfermedad en las nuevas plantaciones. Xac se disemina a corta distancia (dentro de un cuadro) por lluvia y viento (Graham et al., 2004) y a larga distancia (entre países, regiones o entre cuadros) por el material de propagación generalmente asintomático (Graham et al., 2004; Mavrodieva et al., 2004; Hartung et al., 1996; López, 1996). Por ello, se recomienda tener especial cuidado con las plantas de vivero, para evitar esta vía de ingreso de inóculo primario (Goto, 1992). De acuerdo con Hartung et al. (1996) en áreas infestadas la producción de plantas sanas solo es posible si se dispone de técnicas sensibles y específicas que permitan detectar la presencia de Xac. Se han desarrollado diferentes métodos para su detección e identificación siendo los más clásicos el crecimiento del patógeno en diferentes medios de cultivo (Timmer, 1988) y bioensayos en plantas u hojas desprendidas (Koizumi, 1971). En los últimos años se aplicaron técnicas serológicas, principalmente ELISA, utilizando sueros mono y policlonales (Alvarez, 2004; Alvarez et al., 1991; Verniére et al., 1998; Civerolo y Fan, 1982) y PCR con distintos tipos de cebadores (Hartung et al., 1993; Cubero y Graham, 2002; Mavrodieva et al., 2004). Xac es difícil de detectar en bajas concentraciones, como ocurre sobre materiales inertes, árboles asintomáticos o lesiones de menos de tres meses de edad (Mavrodieva et al., 2004). Existen evidencias de la permanencia de Xac por cierto tiempo en tejido asintomático (Goto, 1992; Rossetti et al., 1969) y recientemente se comprobó su capacidad de formar biofilm (Rigano et al., 2007), característica que favorecería su sobrevivencia en bajas concentraciones. El límite de detección logrado mediante la técnica de ELISA es cercano a 104 ufc.mL-1 (Civerolo y Fan, 1982); sensibilidad que fue aumentada a 102 - 103 ufc.mL-1 mediante Inmunofloresencia (IF) (Brlansky et al., 1990) y posibilitó la detección en tejido asintomático (Brlansky et al., 1990; Aubert et al. 1982). A pesar que esta técnica requiere equipos especiales y personal entrenado en la interpretación del material observado, fue utilizada para determinar la diseminación de Xanthomonas campestris pv. citrumelo (Gottwald et al., 1988). La mayor sensibilidad se reporta con los test moleculares PCR y qRT-PCR (PCR cuantitativo en tiempo real) logrando detectar 160 células de Xac por reacción de amplificación por PCR (López et al., 2009).
Esta sensibilidad puede llegar a aumentar a 10 células si los productos de amplificación son detectados por hibridización por Southern-blot (Hartung et al., 1996). La técnica de qRT-PCR recientemente desarrollada, además de la detección e identificación en forma rápida, confiable y sensible de bacterias fitopatógenas, posibilita su cuantificación (Schaad y Frederick, 2002; Weller et al., 2000; Schaad et al., 1999). El uso de PCR en tiempo real para el diagnóstico de cancro cítrico utilizando cebadores diseñados para detectar todas las cepas productoras de cancro, permite llegar al diagnóstico en un tiempo total de cuatro horas con una sensibilidad menor a 10 ufc a partir de lesiones (Mavrodieva et al., 2004). La organización de protección de plantas de Europa y el Mediterráneo (EPPO), luego de un análisis detallado de los métodos propuestos, desarrolló un protocolo para el diagnóstico de Xac en tejido con y sin síntomas que integra más de una metodología (López et al., 2009). Por otro lado, se recomienda que, además de la detección de Xac, se determine su viabilidad; condición que requiere aislar la bacteria en medios de cultivo y producir la enfermedad inoculando plantas indicadoras (López et al., 2009). Todas las especies y variedades de cítricos conocidas son sensibles a Xac, pero las que presentan mayor susceptibilidad son buenas candidatas para su utilización como plantas indicadoras, como es el caso del pomelo, el limón y algunas variedades de naranja dulce (Peltier, 1924).
Con el propósito de brindar una herramienta que facilite la detección de Xac en el material de propagación, este trabajo tuvo como objetivo evaluar la sensibilidad de las técnicas de detección de bacterias para proponer las que presentan el mejor potencial para su ajuste sobre material vegetal asintomático.
Materiales y métodos
Preparación del cultivo
Se utilizó la cepa 49b aislada de fruto de limón y caracterizada por Del Campo et al. (2009). El cultivo madre fue mantenido en glicerol a -70 ºC y cultivado en medio NBXac (23 g.L-1 Nutrient broth, Oxoid, 1g.L-1 glucosa), en agitación (150 rpm) a 28 ºC durante 24 h. A partir de este cultivo se realizó una suspensión DO600 =0.240 correspondiente a 108 ufc.mL-1 (constatado posteriormente por recuento de colonias en placas con medio de cultivo NBXac suplementado con 15 g.L-1 de agar (NBXac agar)). A partir del mismo, se realizaron las diluciones: 10-1, 10-2, 10-3, 10-4, 10-5, ½ 10-5, ¼ 10-5, 1/8 10-5, 10-6 que fueron centrifugadas a 13000 rpm durante 5 min. Los precipitados obtenidos se resuspendieron en 2 mL de tampón fosfato salino (PBS: 8 g.L-1 NaCl, 0,2 g.L -1 KCl, 1,44 g.L -1 Na2HPO4, 0,24 g.L-1 KH2PO4) y 100 µL de cada una de las diluciones fueron sembrados en medio NBXac agar para su cuantificación, y el resto de la suspensión fue utilizado en las distintas técnicas de diagnóstico empleadas.
Detección serológica. Test de ELISA
El test de ELISA indirecto se realizó con el kit AGDIA™, basado en anticuerpos monoclonales que solamente reconocen bacterias que producen el cancro tipo A y el kit producido en la Dirección General de Servicios Agronómicos (DGSSAA) del Ministerio de Ganadería Agricultura y Pesca de Uruguay, basado en anticuerpos policlonales para ELISA-DAS, también específico para cancro tipo A. El procedimiento seguido con los distintos reactivos fue el sugerido por los fabricantes, utilizando microplacas Nunc ™, y analizándose las muestras por duplicado. El desarrollo de color fue detectado y cuantificado en lector de Elisa Dynatech Microscan II, a 405 nm.
Inmunofluorescencia (IF)
Se utilizó el anticuerpo policlonal proveniente del Instituto Biológico de Campinas, Brasil, y como anticuerpo secundario el Anti-rabbit IgG (whole molecule) FITC conjugate de Sigma ®. Sobre portaobjeto alveolado se colocaron 20 mL de la dilución del cultivo a analizar, realizando dos repeticiones por dilución. Se secó a 37 °C y se fijó por inmersión en etanol absoluto durante 10 m.
El anticuerpo específico se diluyó a una concentración de 2 mg.mL-1 y se colocaron 20 mL sobre cada muestra, incubándose a 37 °C durante 60 min. Luego de dos enjuagues de 5 min cada uno por inmersión en PBS, se secó al aire y se colocaron 20 µL del anticuerpo marcado en una concentración de 10 ng.mL-1 en PBS. La incubación se realizó a temperatura ambiente durante 30 min en oscuridad. Se enjuagaron igual que en el paso anterior y se colocaron 20 µL de «antifade» sobre cada muestra para mejorar la estabilidad de la fluorescencia. Se cubrió con el cubreobjetos y se mantuvo en oscuridad a 4 °C hasta su observación en un microscopio Axioplan Z Met de epifluorescencia con objetivo de inmersión 100 X.
Detección Molecular: Reacción en cadena de la polimerasa (PCR)
Se ajustaron las condiciones de amplificación y se compararon los resultados para los siguientes pares de cebadores: P2/P3 y P4/P7 (Hartung et al., 1993) y J-pth1 y J-pth2 (Cubero y Graham, 2002).
Como templado para las amplificaciones en los ensayos de selección de cebadores, se utilizaron diluciones de ADN de Xac extraído por el kit de Sigma™ GenElute Bacterial Genomic DNA Kit. Para los experimentos, la extracción de ADN se realizó utilizando dos procedimientos: a) el método descrito en OEPP/EPPO (Xanthomonas axonopodis pv, 2005). Una colonia fue inoculada en medio NBXac e incubada durante toda la noche a 29 ºC y 150 rpm. Posteriormente, se centrifugó 1,5 mL del cultivo a 10000 rpm durante 10 min y el pellet se resuspendió en 500 µL de tampón de extracción (200 Mm Tris HCl pH 8, 250 mM NaCl, 25 mM EDTA pH 8 0, 5% SDS, 2% PVP).
Luego de una agitación breve se dejó 1 h a temperatura ambiente (18 a 22 ºC) a 150 rpm y se centrifugó a 5000 rpm durante 5 min. Cuatrocientoscincuenta microlitros del sobrenadante fueron transferidos a otro microtubo al que se adicionaron 450 µL de isopropanol obteniéndose una mezcla homogénea, que se dejó a temperatura ambiente durante 1 h y se conservó en freezer a -20 ºC hasta su procesamiento. b) el lisado del cultivo por calor. Se trabajó con las siguientes diluciones en PBS del cultivo original de aproximadamente 108 ufc.mL-1 : 10-1, 10-2, 10-3, 10-4, 10-5, ½ 10-5, ¼ 10-5, 1/8 10-5, 10-6. Cien microlitros de cada una de las diluciones fueron plaqueados en medio NBXac para su cuantificación. Quinientos microlitros de las diluciones originales se centrifugaron a 14000 rpm durante 5 min. El pellet se resuspendió en 100 uL de PBS, que se hirvieron a baño maría por 2 min. Luego, se centrifugaron por 2 min a 13000 rpm y se tomaron 2 uL del sobrenadante para la amplificación por PCR. En las muestras de ADN obtenidas por lisado de las células por calor, se evaluaron los cebadores P2/P3 y P4/P7 (Cubero y Graham, 2002). A partir de este estudio se seleccionaron los cebadores P2/P3 que demostraron mayor sensibilidad, utilizándose para la reacción la composición de la mix y tiempos de ciclado descriptos por Cubero y Graham (2002). La PCR se realizó en un termociclador Quatro TC-40. En cada ensayo se incluyeron testigos positivos para la amplificación de ese fragmento y negativos para controlar bandas inespecíficas o posible contaminación. Los productos de PCR se separaron por electroforesis en gel de agarosa 2% en TAE (Tris-acetato 40mM, EDTA 1mM pH 8) y se visualizaron con luz UV por tinción del gel con bromuro de etidio.
PCR anidada
La secuencia de reacciones de PCR anidado consistió en una amplificación con los cebadores P4/P7 y a continuación la amplificación con P2/P3. Se utilizó ADN purificado con el kit comercial Sigma™ GenElute Bacterial Genomic DNA Kit, en diluciones conteniendo 100 ng, 10ng, 1ng, 100pg, 10pg, 1pg, y 100 fg.
PCR en tiempo real (qRT_PCR)
Para la extracción de ADN, la bacteria se cultivó en medio NAXac hasta una DO600nm = 0,24 que corresponde a 108 ufc.mL-1 . A partir de este cultivo, se realizaron diluciones al décimo y la extracción de ADN se realizó por lisado de la célula mediante calor como fue descripto anteriormente y utilizando los Kits «Genomic DNA extraction Kit» (Fermentas), «EZ_DNA Genomic DNA Isolation Reagent» (Biological Industries™) y «Scorpoclean-Genomic DNA extraction module» (Scorpo Diagnostics™). Cada vez que fue necesario, la concentración de ADN fue evaluada por espectrofotometría con un equipo Nanodrop 1000 o en geles de agarosa, utilizando una concentración conocida del fago l digerido. Se optimizaron las condiciones de amplificación para tres sets de cebadores específicos de regiones conservadas de la familia génica pthA: MV1/MV2, MV3/MV4 y MV5/MV6 según las condiciones de amplificación utilizadas por Mavrodieva et al. (2004) y las del Kit Platinum SyberGreen qPCR Super Mix-UDG (Invitrogen) extraction module» (Scorpo Diagnostics).
Para el ajuste de las curvas de calibración, se partió de concentraciones bajas de ADN (10 a 100 ng.mL-1) y se guardó como curva de referencia aquella que mostró una correlación de 0,99 y alta eficiencia. Para relacionar la concentración de ADN con cantidades de células bacterianas, una alícuota de 100 µL del cultivo original fue cultivada en medio NBXac agar a 28 ºC por 48 h y se realizó el conteo de unidades formadoras de colonias (ufc).
Inoculación en plantas indicadoras
Se utilizaron plantas de naranjo amargo (Citrus aurantum L), pomelo Duncan (Citrus paradisi Macfad), limón (Citrus limonia Osbeck) y mandarina Ponkan (Citrus reticulata Blanco) mantenidos en ambiente controlado a 21-25 ºC y 16 h de fotoperíodo. Teniendo en cuenta el momento de mayor susceptibilidad de la hoja (Gottwald y Graham, 1992), se inocularon en tres plantas de cada una de las especies, dos hojas inmaduras totalmente desarrolladas, por planta. Las hojas fueron inoculadas por infiltración con las alícuotas del cultivo bacteriano resuspendidas en PBS, presionando en la cara abaxial de la hoja con una jeringa sin aguja (Siciliano et al., 2006).
Para el cálculo del volumen inoculado, se infiltraron con agua la mitad de varias hojas con diferentes grados de desarrollo. Con sacabocados de 1 cm2 de área se tomó una muestra de la media hoja sin infiltrar y de la mitad infiltrada. Por diferencia de peso entre tejido infiltrado y no infiltrado de una misma hoja e igual área, se calculó la cantidad de líquido introducido en la infiltración por cm2. Este dato permitió calcular la cantidad de ufc infiltradas en una hoja, conociendo la concentración del inóculo y el área infiltrada. En todas las plantas se inoculó una hoja con PBS como control negativo. La zona inoculada fue marcada con marcador indeleble para el posterior cálculo del área infiltrada. Los síntomas fueron evaluados a partir del tercer día, diariamente hasta 45 días post inoculación (dpi). Las lesiones fueron fotografiadas con una escala que permitió calcular el área infiltrada con el programa GIMP 2.2. Con el conteo de pústulas por área inoculada y la estimación de infiltración de 5 ml.[cm3]-1, se determinó el número de lesiones desarrolladas por mililitro de cultivo. La sensibilidad del test biológico se calculó por la cantidad de pústulas producidas por las diferentes concentraciones de cultivo teniendo en cuenta el volumen de cultivo infiltrado en hoja de las cuatro especies cítricas. Se contabilizaron las pústulas desarrolladas en la zona infiltrada y se midió la superficie infiltrada.
Evaluación de la especificidad de las técnicas moleculares
Como forma de disminuir el riesgo de falsos positivos al aplicar las técnicas moleculares, se analizaron bacterias de diferentes especies del género Xanthomonas (X.pruni, X.fragariae y X.campestris) y dos aislamientos endófitos: Pantoea agglomerans (Erwinia herbícola) y Pseudomonas sp., extraídos de plantas cítricas de un lote comercial e identificados como tales por secuenciación del gen 16S y comparación con secuencias existentes en el banco de datos Gen Bank. Cultivos de estas cepas fueron tratados de igual forma que Xac, y analizados con las mismas técnicas, en las mismas condiciones y en una sola concentración de cultivo, equivalente a aquella de Xac que había resultado inequívocamente positiva por todas las técnicas: 104 ufc.mL-1.
Resultados
La concentración original de células cuantificada en placas con medio de cultivo fueron de 1,8 x 108, 2,1 x 108 y 2,5 x 108 ufc.mL -1 respectivamente para cada una de las repeticiones utilizadas.
Dado que los niveles de sensibilidad alcanzados en cada una de las tres repeticiones del experimento fueron idénticos, se presentan los resultados de la primera repetición. Las concentraciones de bacterias en cada una de las diluciones correspondientes a los datos presentados se detallan en el Cuadro 1.
Sensibilidad de las pruebas serológicas
La determinación serológica con los anticuerpos producidos en la DGSSAA de Uruguay, con el método de ELISA-DAS, permitió detectar Xac hasta en concentraciones 1,8 x 105 ufc.mL-1 y frente a los anticuerpos de AGDIA® utilizados en ELISA-indirecto se aumentó la sensibilidad una unidad logarítmica, alcanzando la detección a 1,8 x 104 ufc.mL-1 (Figura 1). La técnica de inmunofluorescencia aplicada sobre 10 ml de las diluciones descriptas permitió la observación de células bacterianas hasta la última dilución utilizada correspondiente a 180 ufc.mL-1 (Figura 2).
Detección molecular
La sensibilidad obtenida por PCR partiendo de ADN purificado fue menor que utilizando como templado el sobrenadante de las células lisadas por calor. En el segundo caso se obtuvo amplificación hasta 2,2 x 102 ufc.mL-1, equivalente a 22 ufc por reacción, mientras que utilizando ADN purificado se observó amplificación hasta 1 x 103 ufc.mL-1 correspondiendo a 103 ufc por reacción (Figura 3).
El análisis de los diferentes cebadores demostró que la sensibilidad de los cebadores P2/P3 fue de 100 pg, mientras que con los cebadores P4/P7 fue de 10 ng (Figura 4). Sin embargo, las bandas resultantes de la amplificación por PCR a partir de ADN extraído fueron más nítidas y discretas que al utilizar hervido de bacterias enteras, donde se observa una banda más difusa. Utilizando PCR anidado en la primera ronda de amplificación con los primers P4/P7, se obtuvieron bandas en las dos primeras diluciones (100 ng y 10 ng). Para la segunda ronda de amplificación realizada con los cebadores P2/P3, se utilizó como templado 1 uL de las reacciones del primer PCR a partir de la dilución conteniendo 1 ng de ADN que no había manifestado banda visible en el gel de agarosa. En este caso se obtuvo amplificación en los tubos de 1 ng y 100 pg, lo que indica un aumento de la sensibilidad en dos órdenes de magnitud alcanzando a detectar hasta 100 pg de ADN purificado (Figura 5).
Con la técnica de RT_PCR, aplicando el método de extracción de lisado por calor, se logró una sensibilidad de 30 ufc.mL-1, lo que no fue posible al usar kits comerciales.
Sensibilidad en el test biológico
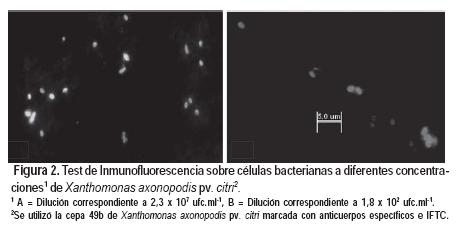
De las cuatro especies indicadoras inoculadas por infiltración, la indicadora que desarrolló síntomas con menor cantidad de bacterias infiltradas fue el naranjo amargo, donde se pudo observar una pústula a la dilución de 1,8 x 102 ufc.mL-1 ( Cuadro 2). De acuerdo al cálculo de volumen de cultivo infiltrado en el área estimada, correspondió a la presencia de dos bacterias en la zona inoculada. Las plantas de pomelo, limón y mandarina Ponkan mostraron la misma sensibilidad (4,5 x 102 ufc.mL-1). La cantidad de ufc necesarias para el desarrollo de al menos una pústula fue coincidente para todas las indicadoras estudiadas en aproximadamente 2 ufc por pústula.
Comparando todas las técnicas evaluadas (Cuadro 3), se observa que la mejor sensibilidad se obtuvo con las técnicas qRT_PCT, inmunoflorescencia y plantas indicadoras. Las menores sensibilidades con las técnicas de ELISA indirecto y PCR a partir de la extracción de ADN.
Especificidad de los métodos
Para ninguno de los test de detección en estudio hubo interferencia de los seis aislamientos filogenéticamente o espacialmente relacionados con Xac utilizados. Además, no se obtuvieron reacciones cruzadas en los test serológicos utilizando anticuerpos policlonales y monoclonales. Los ensayos de PCR no dieron amplificación en estas muestras.
Discusión
Para resolver problemas vinculados al movimiento de material vegetal entre países, programas de certificación y estudios epidemiológicos, se han desarrollado diferentes métodos para la detección de bacterias fitopatógenas, incluída Xac. Con el propósito de disminuir el riesgo local de comercializar material de propagación infectado con Xac, se estudió la sensibilidad de algunas técnicas que detectan diferentes componentes de la célula bacteriana. Los niveles de detección obtenidos en estos ensayos fueron comparables a los citados por otros investigadores (Golmohammadi et al., 2007; Hartung et al., 1996). La IF, el test biológico y qRT_PCR fueron las técnicas que demostraron los mayores niveles de sensibilidad, por lo que se consideran las técnicas más promisorias para el análisis de muestras con poblaciones bajas del patógeno, como es el caso de los tejidos asintomáticos.
La técnica más sensible resultó ser el test biológico, capaz de detectar 2 ufc. Aunque se cita la manifestación de sensibilidades diferentes a Xac entre las especies cítricas utilizadas (Peltier, 1924) en las condiciones del experimento, mostraron igual sensibilidad la mandarina Ponkan (menos sensible) que el pomelo Duncan (muy sensible). La capacidad de desarrollar pústulas de cancro cítrico depende de factores como el estado de los tejidos inoculados, la edad de la hoja (Gottwald y Graham, 1992) y el tiempo en que la hoja permanece húmeda (Dalla Pria et al., 2006), así como de factores ambientales. Estas características que han sido tenidas en cuenta para análisis epidemiológicos de la enfermedad (Dalla Pria et al., 2006; Koizumi, 1971), también deben ser consideradas al evaluar la manifestación de síntomas en plantas de invernadero creciendo bajo condiciones controladas. La aplicación de esta técnica requiere el mantenimiento de las plantas indicadoras en un entorno óptimo de crecimiento que permita disponer de tejido sensible en forma continua, así como un riguroso control de las condiciones de luz, humedad y temperatura. Dentro de las ventajas de la aplicación de este método se encuentra la capacidad de detectar exclusivamente la presencia de células vivas y capaces de inducir síntomas en los tejidos vegetales. La principal limitación de esta técnica parece ser la cantidad de líquido capaz de infiltrase en el tejido de la planta indicadora utilizada que, como se dijo, fue en promedio 5 µl [cm2]-1. Otras limitantes radican en el tiempo que se requiere para la aparición de síntomas y en el alto costo de mantenimiento de plantas en ambientes controlados (Wang et al., 2004). No obstante, es la única técnica que permite tener certeza de la viabilidad de las bacterias detectadas, y no debería descartarse en los test de diagnóstico de Xac (López et al., 2009).
La amplificación por PCR, la segunda en sensibilidad, es altamente específica y rápida, de forma que le permite adaptarse a distintas necesidades comerciales (comercio internacional de cítricos o sus partes, programas de erradicación, etc.) y experimentales. El par de cebadores con el que se obtuvo mayor sensibilidad en el PCR convencional fue el par P2/P3, coincidiendo con Golmohammadi et al. (2007). La secuencia amplificada con estos cebadores, de 222 pb, se encuentra representada en los dos plásmidos portados por Xac (da Silva et al., 2002) lo que significa una duplicación de las secuencias presentes por célula bacteriana (Hartung et al., 1993).
En cuanto a la preparación del templado a utilizar, la variante que incluye el uso de ADN extraído fue menos sensible que las preparaciones a partir de células lisadas por calor. Esta diferencia puede haber sido producida porque los métodos de extracción de ADN no son eficientes cuando las cantidades de ADN son muy bajas. En la medida que con la preparación basada en el lisado por calor de la muestra se alcanza la máxima sensibilidad obtenida en este estudio, tanto para PCR como qRT_PCR, consideramos que es un método ventajoso por su sencillez, rapidez y economía. Una limitante de esta técnica es la sensibilidad de la enzima implicada en la reacción, Taq pol. (Thermus aquaticus polimerase), a la presencia de sustancias inhibidoras como pigmentos, polifenoles y cobre iónico entre otros, lo que da lugar a falsos negativos (Wang et al., 2004). PCR anidada con los pares de cebadores P4/P7 y posteriormente con P2/P3 (Hartung et al., 1996) presentó un alto potencial para ser utilizada con material asintomático ya que, comparada con la PCR común esta técnica demostró ser 50 a 100 veces más sensible en la detección de Xac, usando como templado ADN purificado o células vivas (Hartung et al., 1996). Aunque la qRT_PCR resultó un método rápido, confiable, sensible y con la ventaja de que se minimizó la pérdida de material, el costo actual de los reactivos puede ser una limitante importante en los países no desarrollados. Con el test de ELISA los resultados obtenidos en este trabajo mejoraron el umbral citado por otros autores (Civerolo y Fan, 1982). La IF mostró una buena sensibilidad coincidiendo con los resultados obtenidos por Brlansky et al. (1990). En términos comparativos, la sensibilidad de los test serológicos fue menor que la lograda con las técnicas moleculares, coincidiendo con Wang et al. (2004) donde el test de DIA (Dot Inmunobanding Assay) fue 100 veces menos sensible que la PCR. Una técnica de detección para aplicar sobre material vegetal debe ser específica, muy sensible y robusta, de forma que permita disminuir el riesgo de falsos negativos. El test biológico y la amplificación por PCR mostraron ser las más sensibles para la optimización con muestras de campo sintomáticas y asintomáticas. Es importante tener en cuenta que la cuantificación del cultivo inicial así como de sus diluciones se realizó en base a conteo en medio sólido de las colonias desarrolladas luego de su incubación a 29 ºC por 48 horas sobre medio sólido NBXac. La reciente comprobación de la existencia del estado de viabilidad y no culturabilidad (VBNC) en Xac inducido por cobre (Del Campo et al., 2009), obligaría a utilizar otro método de cuantificación de Xac ya que, de ocurrir este estado en medio de cultivo o en la naturaleza, estaríamos sobreestimando la capacidad de detección de nuestras técnicas.
Como se mencionó, las distintas técnicas detectan distintos componentes de la célula bacteriana y presentan aspectos limitantes. Por ello, consideramos que la evaluación con muestras de campo tanto sintomáticas como asintomáticas debería incluir la visualización de las colonias desarrolladas en medio de cultivo sólido, que permite la recuperación de bacterias vivas y la posibilidad de corroboraciones o estudios posteriores, técnicas de laboratorio de mayor sensibilidad como la PCR y, cuando es posible solventar los costos de implementación y ejecución, se debería incorporar técnicas biológicas como la inoculación en plantas indicadoras o el indexaje en plantas.
Agradecimientos
Los autores agradecen al Programa de Desarrollo Tecnológico (PDT) de la DINACYT por el apoyo financiero obtenido a través del proyecto 77/11. Al personal de apoyo de las estaciones experimentales de INIA_LB, INIA_SG y del IIBCE por la colaboración en el desarrollo de los experimentos.
Bibliografía
Alvarez AM. 2004. Integrated approaches for detection of plant pathogenic bacteria and diagnosis of bacterial disease. Annual Review of Phytopathology, 42: 339 - 366.
Alvarez AM, Benedict AA, Mizumoto CY, Pollard LW, Civerolo EL. 1991. Analysis of Xanthomonas campestris pv. citri and Xanthomonas campestris pv. citrumelo with monoclonal antibodies. Phytopathology, 88: 857 - 865.
Aubert B, Luisetti J, Civerolo EL, Caudet T, Laville E. 1982. Le chancre critique a l’ile de la reunion. Fruits, 37: 705 - 722.
Brlansky RH, Lee RF, Civerolo EL. 1990. Detection of Xanthomonas campestris pv. citrumelo and X. citri from Citrus using membrane entrapment immunofluorescence. Plant Disease, 74: 863 - 868.
Civerolo EL, Fan F. 1982. Xanthomonas campestris pv. citri detection and identification by enzyme-linked immunosorbent assay. Plant Disease, 66: 231 - 236.
Cubero J, Graham JH. 2002. Genetic relationship among worldwide strains of Xanthomonas causing canker in citrus species and design of new primers for their identification by PCR. Applied and Environmental Microbiology, 68: 1257 - 1264.
da Silva ACR, Ferro JA, Reinach FC, Farah CS, Furlan LR, Quaggio RB, Monteiro-Vitorello CB, Van Sluys MA, Almeida NF, Alvez LMC, do Amaral AM, Bertolini MC, Camargo LEA, Camarotte G, Cannavan F, Cardozo J, Chambergo F, Ciapina LP, Cicarelli RMB, Coutinho LL, Cursino-Santos JR, El-Dorry H, Faria JB, Ferreira AJS, Ferreira RCC, Ferro MIT, Formighieri EF, Franco MC, Greggio CC, Gruber A, Katsuyama AM, Kishi LT, Leite RP, Lemos EGM, Lemos MVF, Locali EC, Machado MA, Madeira AMBN, Martinez-Rossi NM, Martins EC, Meidanis J, Menck CFM, Miyaki CY, Moon DH, Moreira LM, Novo MTM, Okura VK, Oliveira MC, Oliveira VR, Pereira HA, Rossi A, Sena JAD, Silva C, de Souza RF, Spinola LAF, Takita MA, Tamura RE, Teixeira EC, Tezza RID, Trindade dos Santos M, Truffi D, Tsai SM, White FF, Setubal JC, Kitajima JP. 2002. Comparison of the genomes of two Xanthomonas pathogens with differing host specificities. Nature, 417: 459 - 463.
Dalla Pria M, Christiano RCS, Furtado EL, Amorin L, Bergamin Filho A. 2006. Effect of temperature and leaf wetness duration on infection of sweet oranges by Asiatic citrus canker. Plant Pathology, 55: 657 -663.
del Campo R, Russi P, Mara P, Mara H, Peyrou M, Ponce de León I, Gaggero C. 2009. Xanthomonas axonopodis pv. citri enters the VBNC state after copper treatment and retains its virulence. Fems Microbiology Letters, 298: 143 - 148.
Golmohammadi M, Cubero J, Peñalver J, Quesada JM, López MM, Llop P. 2007. Diagnosis of Xanthomonas axonopodis pv. citri, causal agent of citrus canker, in commercial fruits isolation and PCR-based methods. Journal of Applied Microbiology, 103: 2309 - 2315.
Goto M. 1992. Citrus canker. En: Kumar J, Chaube HS, Singh US, Mukhopadhyay AN. [Eds.]. Plant diseases of international importance : III diseases of fruit crops. New Jersey : Prentice Hall. pp. 170 - 208.
Gottwald TR, Graham JH. 1992. A Device for precise and nondisruptive stomatal inoculation of leaf tissue with bacterial pathogens. Phytopathology, 82: 930 - 935.
Gottwald TR, Civerolo EL, Garnsey SM, Brlansky RH, Graham JH, Gabriel DW. 1988. Dynamics and spatial distribution of Xanthomonas campestris pv. citri group E in simulated nursery and new grove situation. Plant Disease, 72: 781 – 787.
Graham JH, Gottwald TR, Cubero J, Achor DS. 2004. Xanthomonas axonopodis pv. citri: factors affecting successful eradication of citrus canker. Molecular Plant Pathology, 5: 1 - 15.
Hartung JS, Pruvost OP, Villemot I, Alvarez A. 1996. Rapid and sensitive colorimetric detection of Xanthomonas axonopodis pv. citri by immunocapture and nested-polymerase chain reaction. Phytopathology, 86: 95 - 101.
Hartung JS, Daniel JF, Pruvost OP. 1993. Detection of Xanthomonas campestris pv. citri by the Polymerase Chain Reaction Method. Applied and Environmental Microbiology, 59: 1143 - 1148.
Koizumi M. 1971. A Quantitative Determination Method for Xanthomonas citri by inoculation into detached Citrus Leaves. Bulletin of the Horicultural Research Station Japan Series B., 11: 167 - 182.
López et al., 2009 . Are Molecular Tools Solving the Challenges Posed by Detection of Plant Pathogenic Bacteria and Viruses? Current Issues in Molecular Biology, 11: 13-46.
López MM. 1996. La cancrosis de los cítricos: un peligro potencial para la citricultura española. Levante agrícola, 30(335): 108 - 115.
Mavrodieva V, Levy L, Gabriel DW. 2004. Improved sampling methods for real-time polymerase chain reaction diagnosis of citrus canker from field samples. Phytopathology, 94: 61 - 68.
Peltier GL. 1924. Further studies on the relative susceptibility to citrus canker of different species and hybrids of the Genus citrus, including the wild relatives. Journal of Agricultural Research, 28: 227 - 239.
Rigano LA, Siciliano F, Enrique R, Sandín L, Filippone P, Torres PS, Qüesta J, Dow JM, Castagnaro AP, Vojnov AA, Marano MR. 2007. Biofilm formation, epiphytic fitness, and canker development in Xanthomonas axonopodis pv. citri. Molecular Plant- Microbe Interaction, 20: 1222 - 1230.
Rossetti V, Lasca CC, Namekata T. 1969. Dez anos de observação sobre o cancro cítrico no Estado de São Paulo. Biológico, 35: 45 - 48
Schaad NW, Frederick RD. 2002. Real-time PCR and its application for rapid plant disease diagnostics. Canadian Journal of Plant Pathology, 24: 250 - 258.
Schaad NW, Berthier-Schaad Y, Sechler A, Knorr D. 1999. Detection of Clavibacter michiganesis subsp. sepedonicus in potato tuber by BIO-PCR and an automated real-time fluorescent detectionsystem. Plant Disease., 83: 1095 - 1100.
Siciliano F, Torres P, Sendin L, Bermejo C, Filippone P, Vellice G, Ramallo J, Castagnaro A. 2006. Analysis of the molecular basis of Xanthomonas axonopodis pv. citri pathogenesis in Citrus limon Electronic. Journal of Biotechnology, 9: 199 - 204.
Timmer LW. 1988. Evaluation of bactericides for control of citrus canker in Argentina. Proceedings of the Florida State Horticultural Society, 101: 6 - 9.
Verniére C, Hartung JS, Pruvost OP, Civerolo EL, Alvarez AM, Maestri P, Luisetti J. 1998. Characterization of phenotypically distinct strains of Xanthomonas axonopodis pv. citri from Southwest Asia. European Journal of Plant Pathology, 104: 477 – 487.
Wang ZK, Sun XY, Yin YP, Zhou CY, Xia YX. 2004. Comparision of PCR, DIA and pathogenicity assay for detection of Xanthomonas axonopodis pv. citri, the causal agent of citrus bacterial canker disease. Agricultural Sciences in China, 3: 1 - 6.
Weller SA, Elphinstone JG, Smith NC, Boonham N, Stead DE. 2000. Detection of Ralstonia solanocearum strains with a quantitative, multiplex, real-time, fluorogenic PCR (TaqMan) assay. Applied and Environmental of Microbiology, 66: 2853 - 2858.
Xanthomonas axonopodis pv. citri. 2005. EPPO Bulletin, 35(2): 289 – 294.