Servicios Personalizados
Revista
Articulo
Links relacionados
Compartir
Agrociencia (Uruguay)
versión impresa ISSN 1510-0839versión On-line ISSN 2301-1548
Agrociencia Uruguay vol.15 no.2 Montevideo dic. 2011
Realización de construcciones génicas para el silenciamiento de los virus de citrus Tristeza y Psorosis
Gallino J. Pablo1, Vidal Sabina1, Welin Björn1, Pagliano Gabriela2
1Universidad de la República del Uruguay, Facultad de Ciencias, Iguá 4225, CP11400, Montevideo.
Correo electrónico: jpgallino@gmail.com
2Universidad de la República del Uruguay, Facultad de Agronomía, Av. Garzón 780, CP 12900, Montevideo
Recibido: 31/5/11 Aceptado: 22/7/11
Resumen
El sector citrícola es el de mayor importancia dentro del área hortifrutícola del país, constituyendo el 95% de las exportaciones de frutas y siendo el tercer producto agrícola. Los virus de Tristeza y Psorosis, junto con el viroide de Exocortis y otros viroides asociados a esta enfermedad son los agentes virales y viroidales de mayor incidencia en los cultivos citrícolas de nuestro país. Una estrategia altamente eficaz para el control de enfermedades virales en plantas es la producción de resistencia a través de mecanismos basados en el silenciamiento del ARN. El silenciamiento del ARN es un mecanismo basado en la degradación específica del ARN que es desencadenado por la presencia de ARN de doble hebra dentro de las células. Recientemente se han descrito estrategias para silenciar eficazmente cualquier secuencia de ARN deseada. Utilizando construcciones que se insertan en el genoma de la planta y que promueven la formación de ARN de doble hebra es posible silenciar tanto la expresión de genes endógenos como de secuencias génicas virales. En este trabajo se aislaron secuencias parciales de la cápside de los virus de Tristeza y Psorosis y con ellas se realizaron construcciones génicas que promueven la formación de ARN de doble hebra. Estas construcciones consisten en la inserción de las secuencias virales orientadas en sentido y en antisentido, separadas entre sí por un intrón. Las construcciones obtenidas serán utilizadas para la transformación genética de citrus a efectos de obtener plantas resistentes a estos patógenos.
Palabras clave: CITRUS, VIRUS, SILENCIAMIENTO, CONSTRUCCIONES
Summary
Performing Gene Constructs for the Silencing of Tristeza and Psorosis Viruses in Citrus
The citrus sector is the most important horticultural crop in Uruguay, accounting for the 95% of the fruit exports and being the third agricultural product in order of importance. Citrus Tristeza virus (CTV) and Citrus Psorosis virus (CPsV), together with the viroid for Exocortis and other viroids associated with this disease, are the viral and viroidal agents of highest incidence on citrus crops in our country. A highly effective strategy to control viral diseases is the induction of resistance by means of methods based on RNA silencing. RNA silencing is a mechanism based on the specific degradation of RNA, which is triggered by the presence of double-stranded RNA inside the cells. Recently, strategies to effectively silence any chosen RNA sequence have been described. Using constructs which are inserted into the plant genome and promote the formation of double-stranded RNA, it is possible to silence the expression of both endogenous genes and viral gene sequences. In this study, partial sequences of CTV and CPsV capsids were isolated and used to produce gene contructs that promote the formation of double-stranded RNA. These constructs consist on the insertion of sense and antisense viral sequences, separated by intron. The resulting constructs will be used in the genetic transformation of citrus in order to obtain plants resistant to these pathogens.
Key words: CITRUS, VIRUS, SILENCING, CONSTRUCTS
Introducción
La producción citrícola en Uruguay es un rubro con un fuerte impacto social y económico. Es un sector exportador en expansión, siendo además el de mayor importancia dentro del área hortifrutícola del país (Comisión Honoraria Nacional del Plan Citrícola, 1996). Los virus de Tristeza y Psorosis, junto con el viroide de Exocortis y otros viroides asociados a esta enfermedad son los agentes virales y viroidales de mayor incidencia en los cultivos citrícolas de nuestro país (MGAP, 1981). El virus de la Tristeza de los cítricos es el patógeno viral más importante de este cultivar (Bar-Joseph y Lee, 1989; Lee y Bar-Joseph, 2000). El agente causal es el Citrus Tristeza Virus (CTV), miembro del genero Closterovirus. Las partículas virales son flexuosas y contienen un genoma no segmentado de ARN simple hebra de polaridad positiva que consiste en 19,3 kb con 12 ORFs, que potencialmente codifican para al menos 19 proteínas (Pappu et al., 1994; Vives et al., 1999; Yang et al., 1999).
Los ORFs 7 y 8 codifican para dos proteínas una de 27,4 kDa (p27) y 24,9 kDa (p25). La p25 representa el 95% de la cápside, mientras que el 5% restante está constituido por la p27 (Febres et al., 1996). El agente causal de la Psorosis es el Citrus Psorosis Virus (CPsV) perteneciente al género Ophiovirus (Milne et al., 2000). Se trata de un virus multicomponente con un genoma constituido por tres moléculas de ARN lineales, ARN 1,2 y 3, de polaridad negativa (Milne et al., 2000; García et al., 1997). Para el control de estas y otras enfermedades virales se utiliza la termoterapia y el cultivo de meristemas de forma combinada, con el cual se consigue una reducción de la concentración del virus y/o una aplicación de la zona libre de virus. Los métodos descritos anteriormente son lentos y costosos. Las plantas liberadas a campo a pesar de ser libres de virus, quedan expuestas a posibles contagios por manejo y/o vectores biológicos. En este marco se considera de importancia la incorporación y desarrollo de nuevas estrategias basadas en la ingeniería genética para obtener plantas resistentes a virus por medio del silenciamiento del ARN. Este involucra una serie coordinada de eventos subcelulares que llevan a la terminación post-transcripcional de la expresión génica. Consiste en un mecanismo natural de defensa contra transposones y agentes virales en plantas. Es importante resaltar que existe una conservación muy importante del proceso central involucrado en el silenciamiento génico. La característica más notoria del silenciamiento y que es compartida entre todos los organismos es la formación de pequeños ARN de interferencia, denominados siRNA (small interfering RNA), de 21-26 pb de longitud, que derivan de un ARN de doble cadena. Este es reconocido y cortado por una nucleasa específica del ARN de doble hebra denominada Dicer en Drosophila. Posteriormente los siRNAs se asocian con un complejo denominado RISC (RNA-induced silencing complex) el cual degrada todas las moléculas de ARN que presente homología con los siRNA asociados al mismo. El uso de secuencias virales para producir plantas resistentes a virus es hoy en día una técnica estándar (Reyes et al., 2011; Shimizu et al., 2009; Ludlow et al., 2009; Kreuze et al., 2008; Lin et al., 2011; Di Nicola-Negri et al., 2010; Yan et al., 2010; Kung et al., 2009). Inicialmente se creía que la resistencia provenía de la expresión de proteínas virales (Powell et al., 1986; Lapidot et al., 1993; Brederode et al., 1995).
Se ha demostrado que la producción de ARN de doble cadena es fundamental para el desencadenamiento del silenciamiento génico (Waterhouse et al., 1998; Waterhouse et al., 2001; Matzke et al., 2001; Vance y Vaucheret, 2001). Cuando se transforman plantas con una determinada secuencia nucleotídica se puede inducir el silenciamiento de la misma, mediante la producción de moléculas de ARN de doble cadena, esto ocurre cuando el ADN exógeno es integrado en el genoma como repetidos invertidos (Wang y Waterhouse, 2000). La baja probabilidad de que ocurran estos eventos de integración explicaría el bajo porcentaje de plantas que presentan silenciamiento cuando se las transforma sólo con una secuencia en un mismo sentido, en contraposición a lo observado cuando se utilizan las construcciones diseñadas específicamente para promover la formación de ARN de doble hebra sin importar el sentido de la inserción del ADN exógeno. Estas construcciones génicas reciben el nombre de hpRNA (hairpin RNA) e ihpRNA (intron-spliced hairpin RNA). La eficiencia para desencadenar el silenciamiento utilizando las ihpRNA ha sido determinada en varios experimentos, utilizando diferentes genes, promotores e intrones, siempre observando los más altos porcentajes de individuos silenciados cuando se utilizan construcciones ihpRNA para transformar a las plantas (Wesley et al., 2001). Este trabajo tuvo como objetivo general la realización de construcciones génicas conteniendo secuencias virales con orientación «sentido» y «antisentido», separados por un intrón. Estas construcciones serán utilizadas posteriormente para la transformación genética de citrus mediada por Agrobacterium tumefaciens, esperándose obtener resistencia a la infección de los virus mencionados a través de la activación del mecanismo del silenciamiento génico.
Materiales y métodos
El material vegetal utilizado para el aislamiento del ARN viral consistió en la realización de una selección de plantas indicadoras de cidro Arizona Etrog 861 (C. medica L. Var. etrog Engl.) y naranja dulce Pinneaple (C. sinensis L. Osbeck.) para ambos virus, obtenidos del banco de germoplasma de la Facultad de Agronomía, Universidad de la República.
Extracción de ARN y clonación de las secuencias virales
CTV
Para el virus de Tristeza se utilizó la técnica de inmunocaptura RT-PCR (Olmos et al.,1999), con cebadores que dirigen la amplificación de la secuencia que codifica para la proteína mayoritaria de la cápside (p25). Los oligos utilizados fueron CTrV1h (5´-tcggttccatgaacttac-3´) y CTrV2c (5´-caaattgcgattctgtct-3´). El programa de termociclado fue: 50 min a 37 oC, 92 °C 2 min, seguido de 40 ciclos de: desnaturalización a 92 °C 30 s, hibridación a 45 °C 30 s y extensión a 72 °C 1 min, 72 °C 10 min. El producto de amplificación es de 389 pb.
CpsV
El aislamiento de ARN de CPsV fue llevado a cabo a partir de 1 g de hoja (recién cortada) homogenizada en 3 ml de buffer de extracción (50 mM Tris pH 8,0, 2% SDS, 2% 2-mercaptoetanol). La síntesis de ADN copia fue llevada a cabo en un volumen final de 12,5 ml; 4,5 ml de ARN extraído y 1,25 ml de cada cebador (10 mM) fueron incubados durante 5 min a 65 oC en las siguientes condiciones: 1x buffer RT, dNTPs 200 mM, 40 U RNAsin, 200 U MLV-RT, incubados durante 50 min a 37 oC. Los cebadores utilizados fueron CPsV1h (5´-gcttcctggaaaagctgatg-3´), CPsV2c (5´-tctgtttttgtcaacacactcc-3´) diseñados para obtener un producto de amplificación de 600 pb. La amplificación de la secuencia viral fue realizada en una mezcla de reacción conteniendo: 1x PCR buffer, 200 mM dNTPs, 3 mM MgCl2, 0,5 mM de cada cebador, 5% DMSO, 0,5 ml de ADNc y 1 U de Taq polimerasa (Promega). El programa de PCR utilizado fue: 94 °C, 1 min, 35 ciclos de: 94 °C 30 s, 50 °C 30 s y 72 °C 1 min, 72 °C durante 5 min. Los productos de amplificación fueron ligados en el vector pGEM-T (Promega).
Construcciones para desencadenar el silenciamiento génico
El vector utilizado para generar las construcciones génicas fue el pHAP (cedido por el Dr. Fernando Bravo, INGEBI Buenos Aires) que deriva del vector comercial pUC19 (~2700 pb).
Clonado de las secuencia de CTV en «sentido» y «antisentido»
Para orientar la secuencia del CTV en orientación «sentido» se diseñaron los cebadores CTV.sen.forw (5´-tcggctcgaggaacttacacatc-3´) y CTV.sen.rev (5´-caaattgatatcctgtctacagaaagc-3´), que introducen sitios de corte para las enzimas XhoI y EcoRV, respectivamente. Para orientar la secuencia en orientación anti sentido se diseñaron los cebadores CTV.anti.forw (5´-ccgaattcgcgattctgtctaca-3´) y CTV.anti.rev (5´-tcggttctagaaacttacacatcga-3´), que introducen sitios de corte para las enzimas EcoRI y XbaI respectivamente. La amplificación de las secuencia que sería orientada en «sentido» se llevó a cabo en las siguientes condiciones: 1x buffer de PCR, 0,4 mM de cada dNTP, 0,5 μM de cada cebador (CTV.sen.forw y CTV.sen.rev) 3 mM de MgCl2, 5 U de Taq DNA polimerasa y 10 ng de ADN molde (pGEM-T conteniendo la secuencia viral). El programa de PCR utilizado fue: 94 °C 2 min, seguido de 40 ciclos de: 94 °C 30 s, 52 °C 30 s y 72 °C 1 min, 72 °C durante 10 min. La reacción de ligación del fragmento CTV orientado en «sentido» en el vector pHAP se llevó a cabo con 100 ng de plásmido, 50 ng de inserto, 1x buffer de ligación y 1,5 U de T4 DNA ligasa (Promega), incubado O.N. a 16 °C. La mezcla de ligación fue utilizada para transformar células de E. coli DH5α electrocompetentes utilizando el electroporador Gene Pulser (Bio-Rad). El nuevo plásmido, que contiene el inserto en orientación «sentido» del virus de Tristeza se denominó pMJG1. La amplificación de la secuencia de CTV que sería orientada en «antisentido» se llevó a cabo en una reacción conteniendo: 1x buffer de PCR, 0,4 mM de cada dNTP, 0,5 μM de cada cebador (CTV.anti.forw.EcoRI y CTV.anti.rev.XbaI), 3 mM de MgCl2, 5 U de Taq DNA polimerasa y 10 ng de DNA molde (pGEM-T conteniendo la secuencia cápsidica del CTV). El programa de PCR utilizado fue: 94 °C 2 min, 40 ciclos de: 94 °C 30 s, 56 °C 30 s y 72 °C 1 min, extensión final a 72 °C 10 min. La reacción de ligación del fragmento CTV orientado en «antisentido» se llevó a cabo con 100 ng de plásmido, 70 ng de inserto, 1x buffer de ligación y 1,5 U de T4 DNA ligasa (Promega). El nuevo plásmido, que contiene el inserto de CTV orientado tanto en «sentido» como en «antisentido» se denominó pMJG3.
Clonado de las secuencia de CPsV en «sentido» y «antisentido»
Para orientar la secuencia del CPsV en orientación «sentido» se diseñaron los cebadores CPsV.sen.forw (5´-cttcctcgagaagctgatgacaa-3´) y CPsV.sen.rev (5´-gttgatatcaacacactccatgtcc-3´), que introducen sitios de corte para las enzimas XhoI y EcoRV respectivamente. Para orientar la secuencia en orientación antisentido se diseñaron los cebadores CPsV.anti.forw.EcoRI (5´-tctgaattcgtcaacacactccat-3´) y CPsV.anti.rev.XbaI (5´-tttctagaaaagctgatgacaagcgaa-3´). La amplificación de las secuencia que sería orientada en «sentido» se llevó a cabo en una reacción de amplificación conteniendo: 1x buffer de PCR, 0.4 mM de cada dNTP, 0,5 μM de cada cebador (CPsV.sen.forw y CPsV.sen.rev), 3 mM de MgCl2, 5 U de Taq DNA polimerasa y 10 ng de DNA molde (pGEM-T conteniendo la secuencia viral). El programa de PCR utilizado fue: 94 °C durante 2 min, seguido de 40 ciclos de: desnaturalización a 94 °C durante 30 s, hibridación a 52 °C durante 30 s y extensión a 72 °C durante 1 min, extensión final a 72 °C durante 10 min. La reacción de ligación del fragmento CTV orientado en «sentido» en el vector pHAP se llevó a cabo en 1x buffer de ligación y 1,5 U de T4 DNA ligasa (Promega). El nuevo plásmido, que contiene el inserto en orientación «sentido» del virus de Psorosis se denominó pMJG2. Para la inserción de la secuencia del virus de Psorosis en «antisentido» se realizó la amplifcación de la secuencia en: 1x buffer de PCR, 0,4 mM de cada dNTP, 0,5 μM de cada cebador (CPsV.anti.forw.EcoRI y CPsV.anti.rev.XbaI), 3 mM de MgCl2, 1,5 U de Taq DNA polimerasa y 10 ng de DNA molde (pGEM-T conteniendo la secuencia capsídica del CPsV). El programa de PCR utilizado fue: 94 °C durante 2 min, seguido de 40 ciclos de: 94 °C 30 s, 46 °C 30 s y 72 °C durante 1 min, extensión final a 72 °C 10 min. El nuevo plásmido, que contiene el inserto de CPsV orientado tanto en «sentido» como en «antisentido» se denominó pMJG4.
Construcciones en el vector de transformación
Una vez terminadas las construcciones en el vector pHAP se procedió a removerlas del mismo e insertarlas en el vector binario pKOH200 (Holmström et al., 1996) que será utilizado para la transformación genética de plantas. Para la inserción del cassette del virus de Tristeza en el vector pKOH200 se removió el mismo con la enzima la enzima PstI y se insertó en el sitio SdaI del vector pKOH200. La selección de los transformantes se llevó a cabo en medio LB agar 1,5%, suplementado con streptomicina 25 mg/l. El vector pKOH200 conteniendo el cassette de CTV se denominó pMJG10. Para el cassette de CPsV se removió el cassette del vector pMJG4 con la enzima HindIII y se insertó en el vector pKOH con la enzima HindIII. El vector resultante de la inserción del cassette de CPsV en el vector binario se denominó pMJG11.
Resultados y discusión
Obtención y análisis de secuencias capsídicas de CTV y CpsV
Con el objetivo de obtener secuencias virales se procedió al aislamiento del ARN viral para ser utilizado en reacciones de IC-RT-PCR (inmunocaptura RT-PCR) en el caso de Tristeza y RT-PCR en el caso de Psorosis. Las amplificaciones de mayor eficiencia fueron obtenidas con los cebadores CTrV1h y CTrV2c para CTV, mientras que para CPsV sólo se obtuvieron productos de amplificación con los cebadores CPsV1h y CPsV2c. Por otra parte los tamaños de los productos de amplificación obtenidos para los virus de Tristeza y Psorosis fueron concordantes con lo esperado según los análisis de secuencia (Figura 1). Para CTV se obtuvo un producto de amplificación de 389 pb y para el CPsV se obtuvo producto de amplificación de 600 pb. El fragmento amplificado de la secuencia que codifica para la cápside de Psorosis se extiende desde el nucleótido 654 al 1253, mientras que el fragmento amplificado de la secuencia que codifica para la proteína p25 de la cápside de Tristeza se extiende desde el nucleótido 83 al 471.
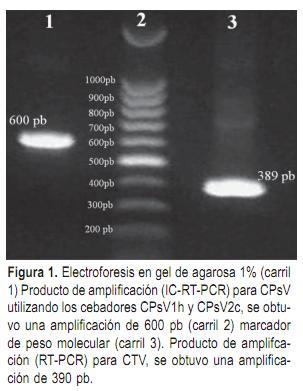
Los productos de RT-PCR fueron clonados en el vector pGEM-T, a partir del cual fue posible realizar la secuenciación, obteniéndose las secuencias nucleotídicas necesarias para proseguir con la estrategia de clonado y para confirmar la identidad de las amplificaciones. La búsqueda de secuencias con homología en el GenBank utilizando el programa BLAST confirmó la identidad de las secuencias dando como resultado un máximo de identidad de 99% y un mínimo de 91% con aislados de CTV, mientras que para CPsV se determinaron un máximo de 87% y un mínimo de 85%. Es necesario más de un 75% de homología para inducir el silenciamiento específico de un determinado gen de forma eficiente (Sijen et al., 1996). Las secuencias obtenidas para los virus en el presente trabajo superan este porcentaje de identidad. Por otra parte, se han obtenido niveles de silenciamiento elevados cuando se utilizan construcciones que promueven la formación de ARN doble hebra de 120 pb (Stoutjesdijk et al., 2002). Las secuencias utilizadas determinan la formación de ARN de doble cadena de aproximadamente 370 pb para CTV y 580 pb para CPsV superando ampliamente la extensión mencionada anteriormente, por lo tanto se espera que el uso de construcciones realizadas con estas secuencias promueva el desencadenamiento efectivo del silenciamiento génico de todas las variantes de las secuencias virales comparadas.
Construcciones génicas para desencadenar el silenciamiento del ARN
La construcción elegida para desencadenar el silenciamiento génico es la llamada ihpRNA. Consiste en la colocación de la secuencia que se desea silenciar, en orientación sentido, separada de la misma secuencia en anti-sentido, por medio de un intrón. Se ha establecido previamente (Wesley et al., 2001) que con este tipo de construcciones se logran los porcentajes más altos de individuos transformantes que presentan silenciamiento génico. Utilizando estos cebadores que introducen sitios de corte para determinadas enzimas de restricción, fue posible primero orientar las secuencias virales en «sentido» en los sitios EcoRV y XhoI del vector pHAP, obteniéndose los vectores denominados pMJG1 y pMJG2 (Figura 2). A partir de estos vectores fue posible insertar las secuencias virales en «antisentido» en los sitios XbaI y EcoRI, obteniéndose los vectores denominados pMJG3 y pMJG4. El intrón presente en el vector pHAP corresponde al del gen ppc1 de S. tuberosum.
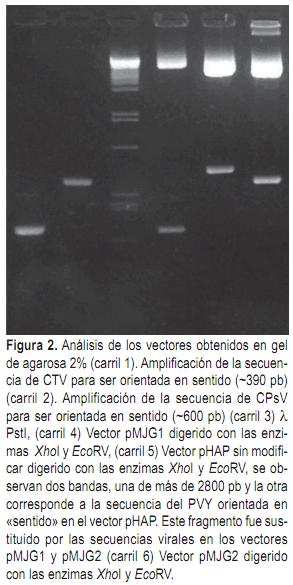
La identidad de la secuencia de los fragmentos clonados en sentido y antisentido fue confirmada por medio de la secuenciación de los mismos.
Obtención de vectores binarios con construcciones que desencadenan el silenciamiento del ARN
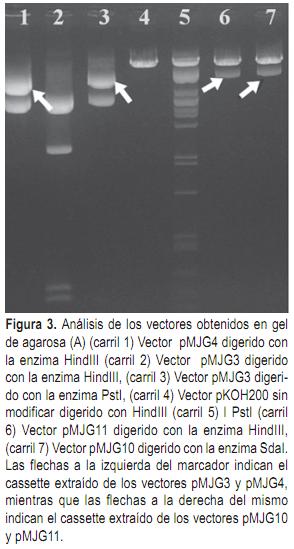
Una vez terminadas las construcciones a partir del vector pHAP y realizados los análisis correspondientes, se procedió a remover los cassettes de los vectores obtenidos (pMJG3 y pMJG4) e insertarlos en el vector binario pKOH200. El cassette de CTV fue removido del vector pMJG3 utilizando la enzima de restricción PstI y clonado en el sitio SdaI del vector pKOH200, el plásmido obtenido se denominó pMJG10 (Figura 3). Para el CPsV se procedió de forma similar, se utilizó la enzima HindIII para remover el cassette del vector pMJG4 y se clonó el mismo en el sitio HindIII del vector binario, de esta manera se obtuvo el plásmido denominado pMJG11.
Análisis bioinformático
La producción de ARN de doble cadena es fundamental para el desencadenamiento del silenciamiento génico. Utilizando las construcciones tipo ihpRNA se obtienen los porcentajes más elevados de transformantes que desencadenan el silenciamiento génico. La funcionalidad del intrón es clave en este tipo de construcciones, por lo que es imprescindible chequear la integridad del mismo. Para ello se llevó a cabo la secuenciación de los extremos del intrón y de las secuencias virales que lo flanquean determinándose que no existe modificación en la secuencia que comprometa la funcionalidad del intrón. A partir de las secuencias obtenidas también se realizó la predicción de la estructura del transcripto de cada construcción, determinándose la formación de horquillas de ARN de doble hebra, tanto para la construcción de CTV como para la construcción de CPsV. Estas horquillas serán reconocidas por el complejo Dicer y procesadas en los siRNAs (21-23nt) que posteriormente determinaran, junto con el complejo RISC, la degradación del ARN que presente homología con los mismos, en este caso el ARN viral que ingrese a la célula.
Conclusiones
Fue posible aislar y clonar parte de las secuencias de los virus de Tristeza y Psorosis, importantes patógenos en muchas variedades de cítricos. Estas secuencias están disponibles para futuros estudios, incluso fueron obtenidas sondas especificas que permiten la detección de estos patógenos. Se dispone de construcciones para promover el silenciamiento en cualquier variedad susceptible a los patógenos mencionados. De esta forma es posible transformar, mediante agrobacterium (cepa EHA-105), tanto variedades de patrones como variedades productoras de fruta si fuera necesario para obtener resistencia. Son las primeras construcciones que se realizan en el país para el desencadenamiento del silenciamiento génico lo cual abre una puerta para la futura utilización de este procedimiento, tanto para estudios genómicos, como para aplicaciones biotecnológicas concretas.
Agradecimientos
Los autores agradecen a la Comisión Sectorial de Investigación Científica (CSIC) por su financiación, a los Ings. Agrs. Beatriz Vignale, Luis Bisio, Alfredo Gravina e Ismael Müller por la provisión de semillas y a todas aquellas personas que han colaborado para llevar adelante esta investigación.
Bibliografía
Bar-Joseph M, Lee RF. 1989. Citrus Tristeza virus. AAB Descriptions of Plant Viruses No 353. Kew Surrey ,England. 7p.
Brederode FT, Taschner PEM, Posthumus E, Bol JF. 1995. Replicase-mediated resistance to alfalfa mosaic virus. Virology, 207: 467-474.
Comisión Honoraria Nacional del Plan Citrícola. 1996. Censo Nacional Citrícola. Montevideo: MGAP. 100p.
Di Nicola-Negri E, Tabaza M, Salandri L, Ilardi V. 2010. Silencing of Plum pox virus 5’UTR/P1 sequence confers resistance to a wide range of PPV strains. Plant Cell Report, 29(12): 1435-1444.
Febres VJ, Ashoulin L, Mawassi M, Frank A, BarJoseph M, Manjunath KL, Lee RF, Niblett CL. 1996. The p27 protein is present at one end of citrus Tristeza virus particles. Phytopathology, 86: 1331-1335.
García ML, Sánchez de la Torre ME, Dal Bo E, Djelouah K, Rouag N, Luisoni E, Milne RG, Grau O. 1997. Detection of citrus Psorosis-ringspot virus using RT-PCR and DAS-ELISA. Plant Pathology, 46: 830-836.
Holmström K-O, Mäntylä E, Welin B, Mandal A, Palva ET. 1996. Drought tolerance in tobacco. Nature, 379: 683-684.
Kreuze JF, Klein IS, Lazaro MU, Chuquiyuri WJ, Morgan GL, Mejia PG, Ghislain M, Valkonen JP . 2008. RNA silencing-mediated resistance to a crinivirus (Closteroviridae) in cultivated sweet potato (Ipomoea batatas L.) and development of sweet potato virus disease following co-infection with a potyvirus. Molecular Plant Patholgy, 9(5): 589-598.
Kung YJ, Bau HJ, Wu YL, Huang CH, Chen TM, Yeh SD. 2009.Generation of transgenic papaya with double resistance to Papaya ringspot virus and Papaya leaf-distortion mosaic virus. Phytopathology, 99(11): 1312-1320.
Lapidot M, Gafny R, Ding B, Wolf S, Lucas WJ, Beachy RN. 1993. A dysfunctional movement protein of tobacco mosaic virus that partially modifies the plasmodesmata and limits virus spread in transgenic plants. Plant Journal. 4: 959-970.
Lee RF, Bar-Joseph M. 2000. Tristeza. In: Compendium of Citrus Diseases, 2nd edn (Ed. Timmer LW, Garnsey SM & Graham JH), pp. 61–63. APS Press, St Paul (US).
Lin CY, Ku HM, Tsai WS, Green SK, Jan FJ. 2011. Resistance to a DNA and a RNA virus in transgenic plants by using a single chimeric transgene construct. Transgenic Research, 20(2): 261-270.
Ludlow EJ, Mouradov A, Spangenberg GC. 2009. Post-transcriptional gene silencing as an efficient tool for engineering resistance to white clover mosaic virus in white clover (Trifolium repens). Journal of Plant Physiology, 166(14): 1557-1567.
Matzke MA, Matzke AJ, Pruss GJ, Vance VB. 2001 RNA-based silencing strategies in plants. Current Opinión on Genetics and Devealopment, 2: 221-227.
MGAP (Ministerio de Ganadería, Agricultura y Pesca). 1981. Enfermedades en plantas hongos superiores y saprofitas en Uruguay. Montevideo: MGAP. Informe técnico Nº9.140p
Milne RG, García ML, Grau O. 2000. Genus Ophiovirus. In: Virus Taxonomy : 7th Report of the International Committe on Taxonomy of Viruses. New York : Academic Press. pp. 627-631.
Olmos A, Cambra M, Esteban O, Gorris MT, Terrada E. 1999. New device and method for capture, reverse transcription and nested PCR in a single closed tube. Nucleic Acids Research., 27: 1564-1565.
Pappu HR, Karasev AV, Anderson EJ, Pappu SS, Hilf ME, Febres VJ, Eckloff RM, McCaffery M, Boyko V, Gowda S, Dolja VV, Koonin EV, Gumpf DJ, Cline KC, Garnsey SM, Dawson WO, Lee RF, Niblett CL. 1994. Nucleotide sequence and organization of eight 3' open reading frames of the citrus Tristeza closterovirus genome. Virology, 199: 35-46.
Powell AP, Nelson RS, De B, Hoffmann N, Rogers SG, Fraley RT, Beachy RN. 1986. Delay of disease development in transgenic plants that express the tobacco mosaic virus coat protein gene. Science, 232: 738-743.
Reyes CA, De Francesco A, Pena EJ, Costa N, Plata MI, Sendin L, Castagnaro AP, Garcia ML. 2011. Resistance to Citrus psorosis virus in transgenic sweet orange plants is triggered by coat protein-RNA silencing. Journal of Biotechnology, 151(1): 151-8.
Shimizu T, Yoshii M, Wei T, Hirochika H, Omura T. 2009. Silencing by RNAi of the gene for Pns12, a viroplasm matrix protein of Rice dwarf virus, results in strong resistance of transgenic rice plants to the virus. Plant Biotechnology Journal, 7(1): 24-32.
Sijen T, Wellink J, Hiriart J-B, van Kammen A. 1996. RNA-mediated virus resistance:role of repeated transgenes and delineation of targeted regions. Plant Cell, 8: 2277-2294.
Stoutjesdijk PA, Singh SP, Liu Q, Hurlstone CJ, Waterhouse PM, Green AG. 2002 hpRNA-Mediated Targeting of the Arabidopsis FAD2 Gene Gives Highly Efficient and Stable Silencing. Plant Physiology, 129: 1723-1731.
Vance VB, Vaucheret H. 2001. RNA silencing in plants defense and counterdefense. Science, 292: 2277-2280.
Vives MC, Rubio L, Lopez C, Navas-Castillo J, Albiach-Marti MR, Dawson WO, Guerri J, Flores R, Moreno P. 1999. The complete genome sequence of the major component of a mild citrus Tristeza virus isolate. Journal of General Virology, 80: 811-816.
Wang MB, Waterhouse PM. 2000 High efficiency silencing of a b-glucuronidase gene in rice is correlated with repetitive transgene structure but is independent of DNA methylation. Plant Molecular Biology, 43: 67-82.
Waterhouse PM, Wang MB, Lough T. 2001. Gene silencing as an adaptive defence against viruses. Nature, 411: 834-842
Waterhouse PM, Graham MW, Wang M-B. 1998. Virus resistance and gene silencing in plants can be induced by simultaneous expression of sense and antisense RNA. PNAS, 95: 13959-13964.
Wesley V, Helliwell C, Smith N, Wang MB, Rouse D, Liu Q, Gooding P, Singh S, Abbott D, Stoutjesdijk P, Robinson SP, Gleave AP, Green AG, Waterhpuse PM. 2001. Construct design for efficient, effective and high throughput gene silencing in plants. Plant Journal., 27: 581-590.
Yan P, Wang S, Shen W, Gao X, Wu J, Zhou P. 2010. Simple construction of chimeric hairpin RNA for virus resistance in plants. Journal of Virological Methods, 166(1-2): 101-5.
Yang Z-N, Mathews DH, Dodds JA, Mirkov TE. 1999. Molecular characterization of an isolate of citrus Tristeza virus that causes severe symptoms in sweet orange. Virus Genes, 19: 131-142.