El descubrimiento de los ARNs no codificantes largos y sus características
Durante varias décadas el dogma central de la biología molecular afirmaba que el ADN era leído a ARN (proceso conocido como transcripción) y a continuación dicha información era “traducida” para la síntesis de proteínas como producto final, para cumplir con los diversos procesos y funciones celulares. Estos ARNs que llevan el mensaje para la síntesis de proteínas son los ARNs mensajeros (ARNm). Se conocían además algunos ARNs que si bien se producían, no eran traducidos luego a proteína. Tal es el caso de los ARNs ribosomales y ARNs de transferencia, que constituyen partes esenciales en la maquinaria de la síntesis proteica.
Con el advenimiento de las nuevas tecnologías de secuenciación a gran escala (secuenciación masiva, o NGS por su sigla en inglés) se ha revelado que, sorprendentemente, la proporción de genoma transcripto a ARN pero que no produce proteínas (ARN no codificante, ya que no contiene el código que especifica su traducción a proteína) es mucho mayor de lo que previamente se estimaba. En humanos, por ejemplo, el ADN no codificante para proteínas alcanza el 98% del genoma, si bien la mayoría es activamente transcripto, es decir, se producen ARNs a partir del mismo1. Esto hace suponer que, dado el gasto energético que ello conlleva para las células, no todo este ARN no codificante sería simplemente “basura” o “ruido transcripcional”, sino que al menos parte de él debería desempeñar alguna función en el organismo.
Estos transcriptos no codificantes son usualmente clasificados de acuerdo a su longitud, en ARNs no codificantes cortos y largos. Ejemplos del primer grupo incluyen ARNs ribosomales (rRNAs), micro ARNs (miRNAs), ARNs de interferencia o silenciamiento (siRNAs), y ARNs de interacción con proteínas PIWI (piRNAs)2. Estos pequeños ARNs, muchos de los cuales cumplen funciones regulatorias y están involucrados en procesos de silenciamiento o apagado de genes3, han sido ampliamente estudiados.
Por su parte, los ARNs no codificantes largos (lncRNAs por su nombre en inglés, long noncoding RNAs) son operacionalmente definidos por oposición a los cortos, como aquellos transcriptos de ARN que presentan al menos 200 nucleótidos de longitud4. Como regla general se asume que estos ARNs no producen proteínas, aunque se ha visto que algunos de ellos podrían dar lugar a pequeños péptidos funcionales5. A medida que se observaron cada vez más ARNs no codificantes en los organismos superiores, y que aumentó el número de lncRNAs identificados (alcanzando más del 65% en todo el genoma humano), los lncRNAs han comenzado a recibir mayor atención6.
Los lncRNAs comparten una serie de características comunes dentro de los vertebrados: presentan pocos exones (es decir, los segmentos que constituyen los ARNs maduros), tienen bajo contenido en las bases nucleotídicas guanina y citosina, están mucho menos conservados en las distintas especies de organismos que los ARNs codificantes, y son mucho menos abundantes que éstos7)(8)(9. Asimismo, se ha evidenciado que la mayor parte de ellos son generados por la ARN polimerasa II, la misma enzima que produce los ARNs mensajeros que luego darán lugar a las proteínas, y por lo tanto la mayoría poseen una cola poliadenilada en su extremo 3’ y un casquete o caperuza metilada en su extremo 5’, que suelen ser características típicas de los ARNs mensajeros10. Por otra parte, también pueden generarse lncRNAs por el procesamiento de otros transcriptos2. Una característica sumamente interesante de los lncRNAs es su patrón de expresión, altamente restringido en tiempo y espacio; es decir, que distintos lncRNAs aparecen en tipos celulares determinados o momentos determinados del desarrollo, con una especificidad mucho mayor de tejido/tipo celular/etapa del desarrollo, que los ARNs codificantes2)(11. A pesar de que en promedio son detectados con no más de una copia por célula, suelen ser abundantes en algunos tipos celulares específicos o incluso en compartimentos sub-celulares particulares, lo que les confiere aún mayor complejidad12.
Funciones y mecanismos de acción de los ARNs no codificantes largos
Si bien el estudio de los lncRNAs ha sido históricamente relegado, en los últimos tiempos se ha comenzado a prestar particular atención a este subgrupo de ARNs, vinculándolos con variados procesos biológicos como diferenciación, proliferación y migración celular, apoptosis, respuesta inmune, desarrollo tisular, etc.4. Investigaciones recientes han mostrado que al menos algunos lncRNAs están asociados a procesos moleculares como regulación de la expresión de genes (a nivel de la transcripción, post-transcripcional, control de actividad de proteínas), modificaciones en la cromatina, organización de dominios nucleares, corte y empalme de ARNs mensajeros (splicing, en inglés), entre otros. Más aún, la alteración en la expresión de algunos lncRNAs se ha relacionado con la generación de tumores; incluso podrían actuar como oncogenes13)(14)(15.
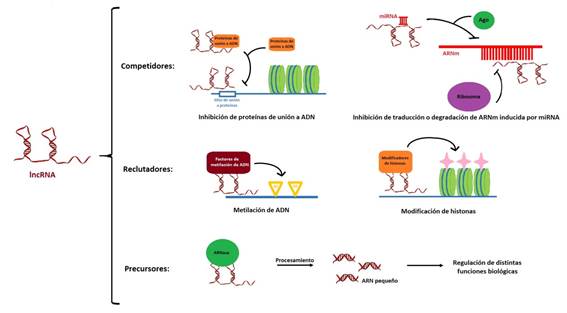
Aunque los roles biológicos y mecanismos de acción de estos ARNs en su mayoría aún se desconocen, existen algunos modelos que intentan explicar su funcionamiento. De modo resumido, como se muestra en la Figura 1, se propone que podrían actuar como: competidores, compitiendo con otras moléculas por el sitio de unión a ADN o proteínas; reclutadores o activadores, activando modificadores del estado de la cromatina (modificaciones epigenéticas); y precursores, siendo procesados y dando lugar a pequeños ARNs, entre otros mecanismos2.
Uno de los lncRNAs cuyo rol ha sido más estudiado es el ARN Xist (en inglés, X-inactive specific transcript). La existencia de dos cromosomas X en las hembras de los mamíferos y sólo uno en los machos, acarrearía el problema de la síntesis del doble de cantidad de ARN y proteína a partir de los genes localizados sobre este cromosoma, en hembras que en machos. La evolución ha solucionado este inconveniente manteniendo activo sólo uno de los cromosomas X de las hembras, en tanto el otro está silenciado. Este mecanismo, que permite balancear la diferencia de expresión derivada del cromosoma X entre ambos sexos, es conocido como “compensación de dosis”16. El lncRNA Xist se transcribe en la vida embrionaria únicamente desde el futuro cromosoma X inactivo, y participa en el silenciamiento de dicho cromosoma en las hembras. El mecanismo de acción de Xist involucra su unión al cromosoma X formando una “nube” a su alrededor que cubre físicamente al cromosoma con su propio transcripto, y reclutando otras proteínas y remodeladores de cromatina para lograr el silenciamiento17. El gen del lncRNA Xist presenta a su vez otro lncRNA antisentido, en la hebra de ADN opuesta, el Tsix, cuya expresión señala al cromosoma X activo. Tsix sirve de regulador de Xist, y es un claro ejemplo de represor antisentido de un gen en la hebra opuesta, que inhibe la expresión de Xist por varios mecanismos18.
Otro modo de acción bastante estudiando es el del lncRNA HOTAIR. Se trata de un ARN no codificante antisentido de los genes Hox, un grupo de genes involucrados en el desarrollo embrionario. El ARN HOTAIR se origina a partir del cluster HoxC (un cluster es un grupo de genes integrantes de una misma familia génica, que a menudo se localizan en una misma región del ADN), y mediante el reclutamiento de ciertos complejos proteicos que estimulan la compactación de la cromatina, silencia la expresión de la región alrededor de los genes HoxD19.
Como ejemplo de precursores, podemos citar al lncRNA H19, que desempeña un rol en la regulación negativa del peso corporal y la proliferación celular. Este ARN constituyó el primer transcripto con impronta identificado. El imprinting o impronta genética es un fenómeno por el cual ciertos genes son expresados o no, según el sexo del progenitor. Ello conduce a la transcripción de una sola copia de un gen autosómico dependiendo del origen parental, en tanto el alelo residente en el cromosoma homólogo está silenciado. H19, que es de expresión materna y está “imprintado” (es decir, silenciado) en el cromosoma paterno, comparte un cluster con el gen Igf2 (factor de crecimiento insulínico tipo 2), que por el contrario, es de expresión paterna y está silenciado en el cromosoma materno. Recientemente se determinó que H19 es precursor de un micro ARN, el miR-675, tanto en humanos como en roedores. Esto parecería indicar que el lncRNA H19 regula la expresión génica mediante un mecanismo basado en la acción de micro ARNs19)(20.
ARNs no codificantes largos y patologías
Se ha sugerido que los lncRNAs estarían asociados a numerosas enfermedades, incluyendo cáncer de diferentes tipos2)(21. En ese sentido, si bien históricamente las causas de la ocurrencia y avance del cáncer se han relacionado principalmente con proteínas, frecuentemente se detecta desregulación de ciertos ARNs no codificantes en pacientes con cáncer22.
Con respecto a los lncRNAs, la expresión aberrante de H19 se ha vinculado desde hace un tiempo a algunos síndromes como los de Beckwith-Wiedemann y Silver-Russell23. Evidencia creciente indica, además, que H19 sería uno de los principales genes involucrados en el desarrollo de numerosos tipos de cáncer, al contribuir a la inestabilidad genómica, aceleración de la proliferación celular y aumento de la resistencia celular al estrés24.
HOTAIR también ha sido vinculado con diversos tipos de cáncer, incluyendo renal, hepático, de mama y esofágico25. Por otra parte, alteraciones en la expresión de H19 y HOTAIR se han asociado con enfermedades cardiovasculares y artritis reumatoidea26.
Algunos trabajos también han relacionado a Xist con tumorigénesis ya que se ha observado la desregulación de su expresión en cáncer de mama y ovario, aunque no puede descartarse que ello sea una consecuencia derivada de la inestabilidad epigenética global en estas patologías18)(27.
Entre los lncRNAs que pueden actuar como competidores, se ha visto que algunos pueden regular los niveles de ARNs mensajeros, compitiendo con micro ARNs. Un ejemplo lo constituye el lncRNA Mir13HG, que está regulado positivamente en un tipo de cáncer de pulmón (NSCLC, por sus siglas en inglés Non Small Cell Lung Cancer)28. Mir13HG es capaz de unirse a miR-214, un micro ARN que ha sido detectado en varios tipos de cáncer, y cuya sobreexpresión inhibe al NSCLC. El lncRNA, al unirse a miR-214 lo secuestra, promoviendo la migración e invasión de células de NSCLC. Dandan y colaboradores28 observaron que silenciando este lncRNA en NSCLC se inhibía la migración, invasión y metástasis de células en dicho cáncer. Por este motivo, lo proponen como un marcador de NSCLC y posible blanco de terapia génica.
Se ha detectado también desregulación de lncRNAs en relación con la fertilidad femenina. En ese sentido, es sabido que una dieta alta en grasas afecta al sistema reproductor. Se ha reportado en ratones que la obesidad afecta la ovulación, el desarrollo ovárico y del ovocito. Además, puede impactar en el embarazo durante la implantación y el desarrollo del embrión, llevando a abortos y preeclampsia, entre otros29. Un trabajo reciente detectó desregulación de lncRNAs en ovarios de hembras de ratón obesas por dieta alta en grasas con respecto al control normal, que incluyó sobreexpresión de numerosos lncRNAs y subexpresión de muchos otros30.
También se ha observado desregulación de numerosos lncRNAs en el síndrome de ovario poliquístico, una de las causas más frecuentes de infertilidad no ovulatoria en hembras, con una prevalencia del 5-10% en el mundo31. Particularmente, el lncRNA HCG26 está sobreexpresado en células de la granulosa de ovarios poliquísticos31. El silenciamiento de este lncRNA conlleva una disminución en la viabilidad celular e inhibición de la progresión del ciclo celular, sugiriendo que la desregulación de este lncRNA contribuiría a la patogénesis del síndrome de ovario poliquístico, aunque los mecanismos moleculares permanecen aún sin aclarar31.
Finalmente mencionaremos la preeclampsia, una de las complicaciones más comunes del embarazo, que causa mortalidad fetal o prenatal. El rol de factores genéticos y epigenéticos se ha sugerido en la patogénesis de la preeclampsia, por lo que diversos estudios se han centrado en el análisis de los lncRNAs. Se han observado patrones aberrantes en su expresión (incluyendo H19, HOTAIR, Meg3, PVT1, Malat1, entre varios otros), aunque los mecanismos subyacentes aún necesitan ser dilucidados32.
ARNs no codificantes largos en el testículo
El testículo se caracteriza por un elevado nivel de diversidad y complejidad transcriptómicas. Exhibe el mayor número de ARNs mensajeros específicos de tejido entre todos los tejidos y órganos estudiados, y una gran complejidad en sus mecanismos de regulación33)(34. Sin embargo, se ha visto que esta diferencia de amplitud en la generación de ARN a partir de ADN en el testículo es mucho más manifiesta aún para regiones intergénicas del ADN (como pseudogenes o elementos transponibles), ARNs no codificantes cortos y lncRNAs, que para los genes que dan lugar a proteínas35. Es decir, que el testículo es el órgano/tejido que presenta los mayores niveles generales de transcripción.
En particular, nos centraremos en los lncRNAs y el hecho de que su presencia es abrumadoramente mayor en el testículo que en otros órganos o tejidos. Es importante además, señalar que este fenómeno se ha observado para los testículos de todas las especies de vertebrados en las que se ha estudiado (Figura 2).
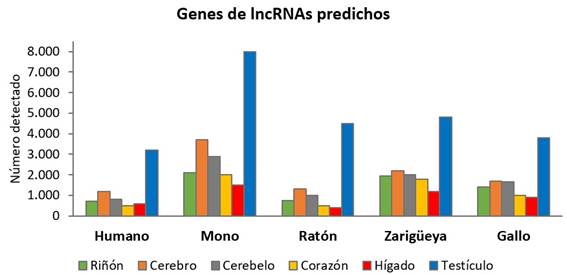
Figura 2: El testículo es el órgano que presenta mayor número de lncRNAs predichos. Se muestran los lncRNAs detectados en seis órganos de cinco especies diferentes: humano, macaco Rhesus (mono), ratón, zarigüeya y gallo. Información extraída de Soumillon et al., 2013 35.
Dado que los lncRNAs en general presentan mayor especificidad de tejido y tipo celular que los ARNs mensajeros, se ha propuesto que su gran abundancia y diversidad en el testículo podría estar sugiriendo su implicancia en el desarrollo de células germinales masculinas14. En el mismo sentido, un estudio realizado en el año 2014 donde se analizó la evolución y conservación de lncRNAs de 11 especies diferentes y también su co-expresión con genes de proteínas, propone que los lncRNAs tendrían funciones asociadas a procesos esenciales, como por ejemplo la espermatogénesis36. Un dato derivado de otro estudio de conservación de lncRNAs testículo-específicos en el ratón, sugiere que la mayoría de los lncRNAs tendrían una baja conservación de secuencia primaria entre especies, y una rápida evolución14.
Diversos estudios han identificado y caracterizado parcialmente un elevado número de lncRNAs en el testículo del ratón14)(35)(37)(38)(39)(40, humano41)(42, u otras especies43)(44. En general estos estudios sugieren que al menos parte de dichos lncRNAs podrían tener un rol regulador o de mantenimiento, afectando al proceso de la espermatogénesis.
De entre los distintos tipos celulares que componen el testículo, la expresión de lncRNAs es más pronunciada en las espermátidas redondas (células posmeióticas), y en los espermatocitos I (células cursando la primera profase meiótica) que en otros tipos de células testiculares35. Nuestro grupo ha observado que la gran mayoría de los lncRNAs testiculares se encuentran en las espermátidas redondas, y los mayores cambios en los patrones de expresión de lncRNAs se detectan en estas células (Trovero et al., manuscrito en revisión). Durante la espermiogénesis (proceso por el cual las espermátidas redondas pasan por una serie de etapas hasta transformarse en espermatozoides), las espermátidas están sujetas a dramáticos cambios en el remodelado de su cromatina, los cuales a su vez requieren de la utilización de diversos mecanismos peculiares de regulación de la expresión génica33)(34. Si bien es probable que muchos de los lncRNAs presentes en las espermátidas representen simplemente “ruido” transcripcional, resulta muy atractiva la hipótesis de que su elevada presencia pudiera estar vinculada, al menos en parte, a la regulación de esos dramáticos cambios cromatínicos y mecanismos de expresión génica, particulares de estas células (Trovero et al., manuscrito en revisión).
Como hemos mencionado más arriba, los lncRNAs revisten un gran interés porque se ha comprobado que contribuyen al silenciamiento génico, inactivación del cromosoma X, establecimiento de la impronta genética, y desarrollo. Sin embargo, muy poco se sabe hasta el momento sobre su biogénesis y función en general, y mucho menos aún de su rol en las células germinales masculinas37. A pesar de ello, existen algunos casos de lncRNAs ya estudiados en detalle, y para los cuales se ha descubierto una función en el testículo y/o la espermatogénesis.
Mrhl (en inglés, Meiotic recombination hot spot locus), es un lncRNA nuclear expresado en el testículo, que regula la vía de señalización Wnt, vía que desempeña un rol esencial en la espermatogénesis de los mamíferos45. HongrES2 regula la maduración espermática, y tendría un rol en la capacitación del espermatozoide46. Tsx (en inglés, Testis specific X-linked) es un lncRNA expresado robustamente en células germinales (específicamente en la primera profase meiótica, en estadio de paquiteno), células madre embrionarias y cerebro, que escapa a la inactivación del cromosoma X, y cuya deleción en ratones aumenta la apoptosis celular durante el paquiteno, derivando en machos con menor tamaño testicular47.
Por otra parte, se han detectado más de 240 lncRNAs específicos de células madre espermatogénicas (SSC), lo que podría indicar su participación en el mantenimiento y auto-renovación de estas células a través de la regulación de proteínas o micro ARNs37. El balance entre la generación y la diferenciación de SSC es indispensable para la espermatogénesis y la fertilidad masculina48. Entre los factores participantes en este proceso, GDNF (en inglés, Glial-Derived Neutotrophic Factor) es un factor de crecimiento de las SSC necesario para su proliferación y mantenimiento. En las espermatogonias tempranas de ratón, incluyendo las SSC, se ha identificado un lncRNA específico (LncRNA033862) que resultó esencial para regular el destino de las SSC. Este lncRNA regula la expresión del gen Gfra1, un receptor del factor GDNF, localizándose dentro de Gfra1 y actuando como antisentido a dicho gen.
Vinculación con patologías testiculares
La infertilidad es un problema de salud mundial que afecta aproximadamente a una de cada 6 ó 7 parejas49. La mitad de estos casos se debe a factores masculinos, aunque el 75% de los pacientes son diagnosticados con causas idiopáticas por desconocerse los mecanismos moleculares que los afectan13. Trabajos recientes han comenzado a revelar un vínculo entre la alteración en la expresión de los patrones de lncRNAs en el testículo, y la infertilidad masculina.
Una de las principales causas de infertilidad en machos es la falta de motilidad de los espermatozoides (astenozoospermia). Un análisis del semen de toros con alta y baja motilidad espermática, dio como resultado una expresión diferencial de lncRNAs: de alrededor de 11.500 lncRNAs identificados en los espermatozoides, unos 2.570 estaban expresados diferencialmente entre ambos grupos, incluyendo 57 lncRNAs sobreexpresados y 2.460 reprimidos en el grupo con baja motilidad50. En general, se considera que muchos de los ARNs presentes en los espermatozoides son una “huella” de eventos ocurridos durante la espermatogénesis, y podrían permitir evaluar el estado de fertilidad del individuo51.
En un estudio reciente sobre el patrón de expresión de lncRNAs en humanos, se identificaron más de 29.000 lncRNAs presentes en esperma y testículo. De entre los ARNs hallados, se identificaron unos 6.300 genes de lncRNAs sobreexpresados y alrededor de 3.400 lncRNAs reprimidos específicamente en muestras de esperma con astenozoospermia, en comparación con muestras normales. Cuando se analizaron los potenciales genes blanco (codificantes para proteínas) de estos lncRNAs, se comprobó un enriquecimiento en genes vinculados a biosíntesis de macromoléculas, espermatogénesis, generación de gametos, metabolismo de ARN, y a vías como la de digestión de proteínas, degradación de ARN, adhesión focal, metabolismo de ácidos grasos y señalización51. Esto sugiere entonces que el metabolismo de proteínas, ácidos grasos y ARN, así como de otras macromoléculas, podrían cumplir roles esenciales en los procesos de espermatogénesis, y su alteración contribuir a la ocurrencia de astenozoospermia.
Por otra parte, un factor de riesgo para la espermatogénesis y la fertilidad masculina es, al igual que para la fertilidad femenina, la obesidad. La obesidad afecta el volumen, la concentración y la motilidad espermática, además de estar también asociada al incremento de daño en el ADN espermático52. Diversos estudios se han focalizado en este problema. Un ejemplo es el trabajo de An y colaboradores52, en el que se identificaron 973 lncRNAs diferencialmente expresados entre muestras de esperma de ratas con obesidad inducida comparadas con ratas normales, revelando una asociación entre los lncRNAs, la reproducción masculina y la obesidad.
Los cánceres de células germinales testiculares (TGCT, del inglés, Testicular Germ Cell Tumours), son el tipo de tumor sólido más frecuente en hombres jóvenes en el mundo desarrollado, y su incidencia ha crecido durante las últimas décadas53. No obstante, la ausencia de modelos de laboratorio apropiados para su estudio, así como las dificultades para el establecimiento de cultivos primarios de células germinales masculinas humanas, representan un obstáculo para el avance del conocimiento acerca de la patogénesis de este tipo de tumores54. Aún así, se ha reportado alteración en el TGCT en algunos genes codificantes, y también en ARNs no codificantes. En particular, se ha detectado expresión del lncRNA Xist en todos los tipos histológicos de TGCT55. Del mismo modo, el lncRNA H19 estaría implicado en este tipo de tumores, dado que los patrones de impronta en la región 5’ de este gen se encuentran borrados por completo en el TGCT; esto marca una diferencia con los tumores de tejidos somáticos, en que la impronta disminuye pero no se borra por completo55.
Los lncRNAs de testículo también pueden estar relacionados con la diabetes mellitus tipo II (TDM2), ya que esta enfermedad tiene efectos estructurales y funcionales sobre el sistema reproductor masculino. La TDM2 paterna influencia las modificaciones epigenéticas durante la espermatogénesis, y esta desregulación epigenética puede ser heredada a las siguientes generaciones por la línea germinal, aumentando el riesgo de TDM2 en la descendencia56. Jiang y colaboradores57 señalaron perfiles de expresión aberrantes de lncRNAs en esperma de ratones diabéticos en comparación con ratones no diabéticos, encontrando unos 7.720 lncRNAs diferencialmente expresados entre ambos grupos (alrededor de 4.310 sobreexpresados y 3.410 regulados negativamente).
Sin embargo, son muy pocos aún los casos en que se ha estudiado la relación de lncRNAs específicos con patologías testiculares. A continuación ilustramos algunos ejemplos de trabajos en los que se vincula a lncRNAs en particular, con el desarrollo de patologías testiculares:
NLC1-C y su vinculación con la azoospermia no obstructiva
Una gran proporción de los casos de infertilidad masculina presenta arresto en la maduración testicular (MA) y azoospermia no obstructiva (NOA), u oligozoospermia severa. En pacientes con NOA se observó un patrón de expresión diferencial de lncRNAs en comparación con pacientes control normales. Se detectaron un total de 757 lncRNAs reprimidos y 475 lncRNAs sobreexpresados en pacientes con MA, y 2.370 lncRNAs reprimidos y 163 lncRNAs sobreexpresados en pacientes con hipoespermatogénesis (es decir, con niveles de espermatogénesis por debajo de lo normal)13.
Entre los lncRNAs regulados negativamente en el grupo de pacientes con NOA, se encuentra el lncRNA NLC1-C (en inglés, NarcoLepsy Candidate-region 1). Se presume que este ARN tendría una función en las etapas tempranas de la espermatogénesis regulando la proliferación o muerte de células germinales, ya que está predominantemente localizado en el citoplasma de las espermatogonias y espermatocitos tempranos en los testículos con espermatogénesis normal. Sin embargo, en los casos de testículos infértiles, el ARN NLC1-C se acumula en el núcleo de estas células13.
NLC1-C es capaz de unirse a proteínas, todas ellas asociadas al procesamiento de ARNs mensajeros inmaduros. Una de estas proteínas es la nucleolina, una proteína nucleolar cuya expresión coincide con la de NLC1-C, encontrándose ambos subexpresados en pacientes con MA y co-localizando en el núcleo celular en los testículos de estos pacientes13.
Por otra parte, el ARN NLC1-C regula micro ARNs, siendo un blanco directo para la unión del miR-320a y el miR-383, y actuando como secuestrador de dichos micro ARNs. Tanto el silenciamiento de NLC1-C como el de la nucleolina incrementan la expresión de ambos micro ARNs, y ambos unidos (NLC1-C y nucleolina) reprimen la expresión de los mismos. La acumulación de NLC1-C en el núcleo de las espermatogonias y espermatocitos primarios, con la consecuente represión de la expresión de miR-320a y miR-383, resulta en una proliferación hiperactiva de células germinales que lleva a la infertilidad13. Además, se ha visto que la acumulación de NLC1-C en el núcleo y su unión a la proteína nucleolina, promoverían también la proliferación celular en el carcinoma testicular embrionario13.
En suma, se propone un modelo para la acción de este lncRNA, que es ilustrado en la Figura 3. En condiciones normales, el lncRNA NLC1-C inhibe a los micro ARNs miR-320a y miR-383, a través de su unión a la nucleolina en el núcleo de las espermatogonias y de los espermatocitos primarios. Al ser exportado al citoplasma junto con los precursores de estos micro ARNs, los mismos son allí procesados en micro ARNs maduros que se unen a NLC1-C para regular la espermatogénesis. Sin embargo, cuando el NLC1-C está regulado negativamente en el citoplasma (no está claro aún si lo que falla es la exportación desde el núcleo al citoplasma) y es acumulado en el núcleo reprimiendo a los transcriptos precursores de miR-320a y miR-383, lleva a la hiperactiva proliferación de las células germinales mencionada mediante su unión a la nucleolina, lo que en última instancia deriva en infertilidad masculina13.
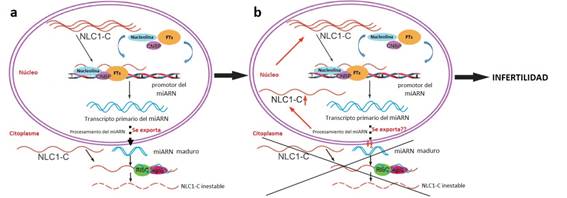
Figura 3: Modelo de la desregulación del lncRNA NLC1-C durante la espermatogénesis, que deriva en azoospermia no obstructiva e infertilidad. (a) En el testículo normal, NLC1-C es exportado al citoplasma junto a los precursores de los micro ARNs, que son procesados en el citoplasma. (b) Bajo desregulación de NLC1-C, éste se acumula en el núcleo, y junto a la nucleolina, inhibiría la producción de los precursores de los micro ARNs. Tomado y modificado de Lü et al., 201513.
SPRY4-IT1 y los tumores de células germinales testiculares
Un estudio realizado empleando líneas celulares de carcinoma embrionario derivado de tejido metastásico de TGCT, detectó sobreexpresión del gen codificante para la proteína SPRY4 y de su lncRNA intrónico SPRY4-IT1 en muestras de TGCT en comparación con muestras de testículo normal. Al ser un lncRNA intrónico, el ARN SPRY4-IT1 se produce a partir de una región del gen SPRY4 que no produce proteína. Los patrones de expresión del gen codificante y del lncRNA difirieron notablemente entre los distintos tipos de TGCT: mientras la expresión del gen codificante era mayor en los tumores de saco vitelino, carcinoma embrionario y coriocarcinoma, los niveles de expresión del lncRNA eran mayores en teratomas. Las diferencias en el patrón de expresión entre el gen codificante y el lncRNA, junto con el hecho de que la disrupción de uno de ellos no alteraba la expresión del otro, que el lncRNA presentaba mayor sensibilidad a factores de crecimiento que el gen codificante, y que la degradación de ambos transcriptos estaba regulada independientemente, sugieren que ambos funcionarían como transcriptos independientes54.
Un descenso en los niveles de expresión de SPRY4 y de SPRY4-IT1 resultó en disminución de la viabilidad, proliferación, migración y capacidad de invasión celular. Por otra parte, la supresión de la expresión tanto del gen codificante como del lncRNA mostró un efecto inhibitorio sobre las vías mediadas por receptores de tirosín-kinasas (RTK), al inhibirse principalmente la fosforilación de la kinasa Akt y, en menor medida, la fosforilación de las kinasas ERK1/2 (esta última fue dependiente del producto del gen codificante pero no del lncRNA) (54). Estos factores son parte de las cascadas de señalización MAPK / ERK y PI3K/ Akt, involucradas en la homeostasis del crecimiento y diferenciación celular.
En conclusión, los resultados sugieren que tanto el gen codificante SPRY4 como su lncRNA SPRY4-IT1 actuarían como oncogenes en el TGCT, y que el bloqueo de la expresión de ambos inhibe el crecimiento de TGCT, al inhibir la activación de la vía PI3 / Akt (Figura 4). De todos modos, aún restan muchos estudios para comprender mejor los mecanismos de acción de estos genes y, en particular, del lncRNA SPRY4-IT1, en la patogénesis del cáncer testicular.
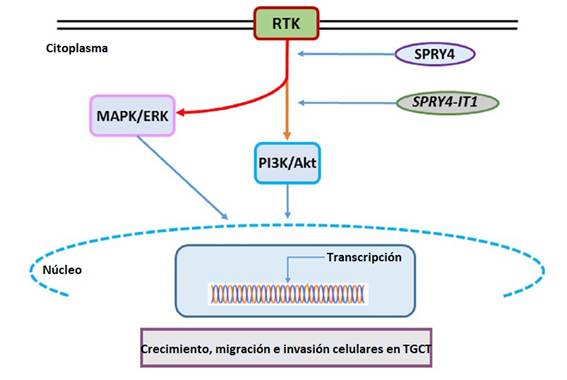
Figura 4: Modelo de activación de las vías MAPK/ERK y PI3K/Akt por SPRY4 y el lncRNA SPRY4-IT1 en células con TGCT. RTK: receptores de tirosín-kinasas. Tomado y modificado de Das et al., 2018 54.
Malat1 y su participación en la isquemia testicular
La torsión testicular afecta principalmente a hombres adolescentes y adultos, e involucra una torsión aberrante del cordón espermático que conlleva isquemia. Si esta falta de irrigación sanguínea es prolongada puede ocasionar daños irreparables, por lo que debe ser tratada rápidamente. Pero a su vez la detorsión puede provocar daño por reperfusión que puede ser aún más grave, lo que se conoce como daño por reperfusión de isquemia (en inglés, Ischemia-Reperfusion Injury o IRI)58.
Se estudió el rol del lncRNA Malat1 en IRI testicular en ratones, observándose que su expresión se incrementa abruptamente luego de una hora de isquemia y 8 horas de reperfusión. Además, se comprobó que la sobreexpresión de este lncRNA inhibía notablemente la viabilidad y proliferación celular, promoviendo la apoptosis en las células testiculares espermatogénicas58.
Como hemos visto, los lncRNAs pueden unirse a micro ARNs, modulando a su vez la represión de los blancos de los micro ARNs. El micro ARN miR-214 está inversamente expresado en IRI testicular con respecto a Malat1, y la sobreexpresión de Malat 1 reduce significativamente la expresión de miR-214, aunque este efecto no es recíproco. Ello indica que miR-214 es un blanco inhibitorio del lncRNA Malat1 durante la progresión del IRI testicular58. A su vez, la sobreexpresión de Malat1 promueve la expresión del gen TRPV4 (en inglés, Transient Receptor Potential Vanilloid 4), un canal catiónico no selectivo cuya excesiva activación se ha visto en IRIs en miocardio, cerebro y pulmón; sin embargo, la presencia de miR-214 interrumpe este efecto. Se ha demostrado que la inducción de miR-214 inhibe la expresión de TRPV4, en tanto la represión del micro ARN aumenta la expresión del gen. En conjunto, la regulación de TRPV4 por Malat1 requiere de la actividad de mirR-21458.
En resumen, la sobreexpresión de Malat1 es una característica molecular de la IRI testicular, que aparece asociada positivamente a la muerte celular inducida en IRI testicular, y negativamente a la proliferación celular a diferentes tiempos de reperfusión. Se identificó al micro ARN miR-214 como blanco de Malat1, y éste último inhibiría la expresión de miR-214. Por el contrario, el gen TRPV4 sería un blanco inhibitorio del miR-214 que actúa como sensor para la IRI testicular58.
LncRNAs desregulados y síndrome de Klinefelter
El síndrome de Klinefelter (SK) es un fenómeno común en hombres, con una frecuencia aproximada de 1:60059. Este síndrome se caracteriza por la presencia de al menos un cromosoma X extra, a consecuencia de la no disyunción del par de cromosomas X durante alguna de las divisiones meióticas incluidas en el proceso de formación de los gametos masculinos. El 90 % de los casos del SK no corresponde a mosaicos sino que se trata, por ejemplo, de individuos con cariotipo XXY, siendo éste el cariotipo mayoritario. Si bien los hombres con SK pueden presentar focos de espermatogénesis en sus testículos, el 90% de los casos no-mosaico presenta oligozoospermia severa o azoospermia completa60.
Recientemente se publicaron dos trabajos que relacionan al SK con lncRNAs. Winge y colaboradores61 analizaron testículos fetales con SK y normales, obtenidos de abortos. Observaron una pérdida significativa de células germinales en los testículos fetales con SK en comparación con los normales, que es una característica propia de los testículos adultos con SK. Los autores sugieren por lo tanto que esta pérdida comenzaría en el feto, por una falla en la diferenciación de los gonocitos hacia pre-espermatogonias. Al analizar el transcriptoma (es decir, todo el ARN presente) en los testículos fetales con SK, y compararlos con testículos normales, encontraron que entre los transcriptos diferenciales entre ambas condiciones había un enriquecimiento de lncRNAs ubicados en regiones intergénicas (o sea, regiones que no contienen genes codificantes para proteínas) en el genoma de los testículos con SK. En menor medida, también observaron un enriquecimiento en lncRNAs antisentido (transcriptos a partir de la hebra de ADN opuesta a un gen que da lugar a una proteína) y lncRNAs sentido-intrónicos (en la misma hebra que un gen de una proteína, pero ubicados en la región intrónica del mismo). Sólo 2 de los 37 lncRNAs intergénicos expresados diferencialmente en el testículo con SK correspondieron a la transcripción de secuencias del cromosoma X. Uno de ellos es el lncRNA Xist, un resultado esperable ya que como fue explicado anteriormente, Xist colabora en la inactivación del X, y se genera a partir del mismo; en consecuencia, si hay un X supernumerario, no es extraño encontrar sobreexpresión de Xist. Este artículo propone que la pérdida de las células germinales en el feto de los pacientes con SK podría vincularse con la expresión aberrante de lncRNAs61.
El segundo artículo publicado recientemente en relación a SK y lncRNAs se centra en el lncRNA GAS5 (en inglés, Growth Arrest-Specific 5). En este trabajo se compararon testículos con SK y testículos normales de igual edad, encontrándose 4.448 genes expresados diferencialmente en SK, entre los cuales el locus GAS5 estaba sobreexpresado62. Este lncRNA está asociado a diversos procesos biológicos como la represión de la acción de glucocorticoides, enfermedades inflamatorias y autoinmunes, apoptosis vascular, ateriosclerosis, y proliferación de células madre germinales femeninas62. Debido a su relación con los eventos de crecimiento, proliferación y desarrollo celular, señalización, e interacción célula-célula, se sugiere un posible rol para GAS5 en la pérdida de células germinales en los testículos de pacientes con SK. De todos modos, si realmente su sobreexpresión es causa de esta característica fenotípica en la enfermedad, es algo que aún queda por investigar62.
Los antígenos “cancer-testis”
Un caso diferente aunque muy interesante, es el de los “cancer-testis antigens”. Si bien en sentido estricto no deberíamos incluirlos en esta revisión dado que no se encuentran involucrados en patologías testiculares, elegimos mencionarlos por lo curioso de los mismos. Se trata de un grupo de genes de expresión normal exclusivamente en el testículo, y cuya expresión en otros tejidos está asociada al cáncer, hecho del cual deriva su nombre.
Dentro de este grupo de los cancer-testis antigens se incluyen algunos lncRNAs. Un ejemplo lo constituye el lncRNA DBF4B, una isoforma no codificante del gen DBF4B, de expresión diferencial de espermatogénesis pero inhibida en espermatocitos, y que es regulada positivamente en el cáncer gástrico. Otro caso es el del lncRNA antisentido DLG1, que es específico del testículo y se encuentra sobreexpresado normalmente en los espermatocitos. Sin embargo, su expresión se detecta también en carcinoma escamoso, melanoma maligno, y enfermedad de Hodgkin. El lncRNA antisentido KIRREL3-2 es también diferencial de espermatogénesis y sobreexpresado normalmente en los espermatocitos, con un posible rol en la meiosis. Su presencia se ha detectado también en linfoma, enfermedad de Hodgkin, adenocarcinoma papilar, y melanoma maligno63.
En 2018 Cheng y colaboradores64 relacionaron al lncRNA THOR (en inglés, Testis-asocciated Highly conserved Oncogenic long non-coding RNA) con el carcinoma hepatocelular (HCC), ya que lo detectaron altamente expresado en muestras de pacientes con dicha patología. Observaron inhibición del crecimiento y de la metástasis de HCC mediante la depleción de THOR.
El lncRNA TDRG1 (en inglés, Testis Development-Related Gene 1) mostró elevada expresión en tejidos de cáncer epitelial de ovario (EOC), observándose que la regulación negativa de este lncRNA suprimía la proliferación, migración e invasión de células de EOC. Se ha propuesto que TDGR1 actuaría induciendo el EOC mediante la reducción de la expresión del micro ARN miR-93 y sus genes blanco, por lo que no sólo podría considerarse un marcador de EOC, sino también un posible blanco terapéutico65.
Perspectivas y posibles aplicaciones de los ARNs no codificantes largos
Con el surgimiento de las nuevas tecnologías de secuenciación, ha quedado en claro que la proporción de ADN que codifica para proteínas representa el porcentaje más pequeño del genoma, y que la inmensa mayoría corresponde a ARNs no codificantes. Dentro de éstos, un grupo mayoritario lo conforman los lncRNAs, cuyo descubrimiento ha cambiado dramáticamente la visión sobre la genética. Últimamente se les ha comenzado a prestar particular atención ya que, como hemos visto, se ha acumulado evidencia creciente que demuestra su vínculo con diversos procesos biológicos, principalmente con funciones regulatorias, incluida la espermatogénesis (recordemos que el testículo es el tejido que expresa el mayor número de lncRNAs). En este trabajo, hemos revisado algunos de los diferentes estudios que los han relacionado también a diversas patologías. En este sentido, se ha propuesto a los lncRNAs como posibles biomarcadores para las diferentes afecciones, y también como posibles blancos de terapias génicas.
En particular para el caso del cáncer, el método más común de obtención de muestras tumorales ha sido la biopsia de tejido, que conlleva riesgo y es muy dolorosa para el paciente, además de económicamente costosa. Existe una nueva metodología de diagnóstico, la “biopsia líquida”, que es menos invasiva, reduce los riesgos, y, dependiendo del tipo de cáncer en cuestión, podría ser tomada de sangre, orina, saliva, entre otros, permitiendo detectar biomarcadores circulantes, tales como proteínas y ácidos nucleicos(66). A pesar de este avance en los métodos diagnósticos, las pruebas basadas en detección de proteínas no son suficientemente selectivas y sensibles. Por otra parte, el análisis en el ADN de mutaciones en genes de cáncer no es específico de tejido, por lo que se necesitan análisis adicionales. En consecuencia, se está trabajando en la búsqueda de biomarcadores alternativos para el diagnóstico, que sean confiables y eficientes sobre todo en etapas tempranas de la tumorigénesis66.
El transcriptoma circulante representa una rica fuente de biomarcadores para el cáncer, incluyendo tanto ARNs codificantes como no codificantes. Entre estos últimos, los micro ARNs han sido estudiados exhaustivamente como potenciales biomarcadores. Sin embargo, en este último tiempo también ha comenzado a ponerse interés en otros tipos de ARNs no codificantes, como los lncRNAs67.
Los lncRNAs pueden funcionar como oncogenes (causan el crecimiento descontrolado de células normales y las convierten en tumorales) o como supresores tumorales (protegen a las células de transformarse en células cancerosas). La transcripción aberrante de los mismos se ha confirmado como un predictor confiable de cáncer. Además, en múltiples tipos de cáncer se han encontrado lncRNAs circulantes en plasma, suero, orina, así como también en exosomas67.
El PCA3 (en inglés, Prostate Cancer Antigen 3) es un lncRNA específico de próstata que se sobreexpresa en el cáncer de próstata, y es utilizado en una prueba diagnóstica de esta enfermedad donde se mide su concentración y la relación con otro biomarcador, en muestras de orina68. MALAT1 se ha estudiado exhaustivamente como lncRNA circulante, habiéndose encontrado en plasma y suero de pacientes con cáncer de próstata, y en exosomas de pacientes con otros tipos de cáncer. Asimismo se han identificado otros lncRNAs circulantes en pacientes con cáncer, incluyendo HOTAIR y GAS5, entre otros67.
Además de su potencial uso como biomarcadores, los lncRNAs podrían ser empleados como blanco de terapias, debido a su gran especificidad de tejido, célula o tumor69. Esto podría llevarse a cabo mediante degradación postranscripcional del ARN, modulación de los lncRNAs por bloqueo de su promotor o utilizando técnicas de edición genómica efectivas y sin blancos inespecíficos como lo es la tecnología CRISPR, o mediante pérdida de función del lncRNA ya sea con inhibidores de interacciones ARN-proteínas o impidiendo la formación de estructuras secundarias en el ARN69)(70. A modo de ejemplo, un reporte reciente indica que la inhibición del lncRNA H19 podría constituir una terapia efectiva para el tratamiento del cáncer de páncreas71.
A pesar de todos los esfuerzos puestos en el estudio de los lncRNAs y en la elucidación de sus funciones, el mundo de los lncRNAs está aún en sus inicios y mucho queda aún por hacer para lograr identificar y describir sus mecanismos moleculares de acción. Sin dudas, los próximos años marcarán importantes avances en el conocimiento de este novedoso grupo de ARNs, y en su eventual utilidad para el diagnóstico y la terapia. Dada la gran abundancia de los lncRNAs a nivel testicular, estos avances serán especialmente relevantes para el mejor conocimiento de los eventos moleculares subyacentes al complejo proceso de la espermatogénesis, así como para el diagnóstico y tratamiento de patologías como la infertilidad y el cáncer testicular.