Introducción y antecedentes
Las Periodontitis Agresivas son enfermedades periodontales destructivas que pueden afectar la salud de la dentición primaria o permanente y en un período corto de tiempo pueden llevar a la pérdida de los tejidos de soporte del diente. El término Periodontitis Agresiva fue aprobado en el Workshop for the Classification of the Periodontal Diseases and Conditions organizado por la American Academy of Periodontology (AAP) en 1999 para definir un grupo de enfermedades periodontales destructivas con una progresión rápida, clasificación vigente al momento de la realización de este trabajo 2-4.
Las Periodontitis Agresivas pueden presentarse en forma localizada Periodontitis Agresiva Localizada (PAL) o generalizada Periodontitis Agresiva Generalizada (PAG) de acuerdo a la extensión de la destrucción periodontal 1.
Según la Organización Mundial de la Salud (OMS) las enfermedades periodontales graves, que pueden desembocar en la pérdida de dientes, afectan a un 15%-20% de los adultos de edad media (35-44 años) 5. En cuanto a los cuadros agresivos, Albandar menciona que la prevalencia de la Periodontitis Agresiva es variable si se toma en cuenta diferentes localizaciones, si bien muchos de estos estudios utilizaron variables y metodologías diferentes. La prevalencia de la Periodontitis Agresiva Localizada varía considerablemente entre continentes y las diferencias entre razas/etnias parece ser un factor importante. Diferentes estudios mostraron en forma consistente que la Periodontitis Agresiva es más prevalente en África y en poblaciones afrodescendientes y menos prevalente en población caucásica de Europa y Norteamérica. Entre niños y adultos jóvenes la prevalencia es superior en el grupo etario mayor que en los más jóvenes 6. En Europa la prevalencia de la Periodontitis Agresiva es baja entre 0.1% y 0.2% y prevalencias más altas entre 3 y 10% en Brasil y Estados Unidos 7. En América Latina, estudios muestran que la prevalencia de la Periodontitis Agresiva es mayor en esta región que en los países industrializados. El rango de prevalencia varía entre 0.3% a 4.5% siendo la forma localizada la menos prevalente 8. A nivel mundial, la prevalencia de PAL es menor a 1%, la de PAG es de 0.13% 9-10.
Los cuadros de Periodontitis Agresivas responden a un origen multifactorial como es la respuesta del hospedero, factores genéticos y la microbiota subgingival 6,9,11.
Algunas de las especies bacterianas patógenas que han sido asociadas con el desarrollo de la Periodontitis Agresiva son: Aggregatibacter actinomycetemcomitans (Aa)2,12 y Porphyromonas gingivalis (Pg)13 siendo el primero particularmente importante en cuadros localizados 14-15 y el segundo en generalizados 15-16) pero también se han relacionado otro grupo de periodontopatógenos como Tannerella forsythia (Tf), Prevotella intermedia (Pi)1-15 y Fusobacterium nucleatum (Fn)15.
En Uruguay se han realizado estudios utilizando metodologías de identificación bacteriológicas tradicionales pero no existen hasta el momento estudios en relación a los niveles de prevalencia simultánea de los patógenos periodontales mencionados anteriormente en los cuadros de Periodontitis Agresiva. Por otra parte, las técnicas bacteriológicas tradicionales presentan ciertas limitaciones como el mantenimiento de la viabilidad de la muestra, el tiempo de desarrollo bacteriano (de 7 a 15 días) así como la dificultad del crecimiento bacteriano ya que responden a microorganismos que requieren nutrientes particulares y son en su mayoría anaerobios estrictos. Además el reconocimiento bacteriano implica la realización de pruebas adicionales, como pruebas bioquímicas, para la identificación de la especie 17.
Con la introducción de los métodos de estudio moleculares como la reacción en cadena de la polimerasa (PCR) se ha podido ampliar y simplificar los estudios de la composición de la microbiota oral de los pacientes con patologías periodontales, mejorando la sensibilidad, especificidad y eficiencia 18.
En nuestro trabajo se analizó la presencia de cinco patógenos periodontales: Aggregatibacter actinomycetemcomitans, Porphyromonas gingivalis, Prevotella intermedia, Tannerella forsyhtia y Fusobacterium nucleatum en las bolsas periodontales de pacientes uruguayos de la Clínica de Periodoncia de la Facultad de Odontología de la Universidad de la República de Uruguay. Las técnicas de identificación microbiológica incluyeron técnicas bacteriológicas tradicionales así como técnicas de identificación del material genético presente a través de PCR 17.
Con este estudio se pudo identificar qué microorganismos están presentes y son más prevalentes en los cuadros de Periodontitis Agresiva en Uruguay tanto en los cuadros generalizados como localizados y determinar si se mantienen los niveles de prevalencia que aparecen en otras localizaciones geográficas.
Además, permitió correlacionar las técnicas bacteriológicas tradicionales con las técnicas moleculares.
Metodología de trabajo
Se seleccionaron 50 pacientes de la Clínica de Periodoncia de la Facultad de Odontología con cuadros de Periodontitis Agresiva, sin previo tratamiento periodontal y que hayan firmado el consentimiento informado. Se exceptuaron pacientes con Diabetes, Artritis, Colitis Ulcerativas, VIH, Cáncer y Patología Cardiovascular, mujeres embarazadas y aquellos que recibieron tres meses antes tratamientos con antibióticos y/o antiinflamatorios. El comité de ética de la Facultad de Odontología UDELAR, aprobó el diseño del estudio basándose en la normativa MERCOSUR y la Declaración de Helsinki sobre la experimentación en la cual participan seres humanos.
Las muestras se obtuvieron removiendo la placa supragingival evitando el sangrado usando gasa estéril. La toma se realizó introduciendo puntas estériles de papel medianas (Nº25) en lo más profundo de la bolsa y dejándolas durante 15 segundos. Luego de esto, se colocaron en 1.5 ml de medio reducido RTF (Reduced Transport Fluid) 18. Cada muestra constó de 8 puntas de papel de 4 sitios seleccionados en cada cuadrante.
El diagnóstico bacteriológico a través de metodología tradicional se realizó para A. actinomycetemcomitans y para los anaerobios pigmentados P. gingivalis y P. intermedia. La identificación por PCR se realizó además de los anteriormente mencionados para T. forsythia y F. nucleatum. Las muestras se procesaron en el laboratorio de Microbiología de la Facultad de Odontología realizando la agitación rigurosa durante 45 a 60 segundos y posteriormente realizando diluciones seriadas en RTF. Se tomaron 100 µl de la muestra colocándolos en un tubo Eppendorf con otros 900 µl de RTF logrando una dilución de 1:10. De ésta se sembraron 100 µl expandiendo la muestra con una varilla de vidrio sobre una placa de TSVB: tripticasa soya, bacitracina (75 µg/ml), vancomicina (5.0 µg/ml), 10 ml de suero de caballo (10%) para el aislamiento de A. actinomycetemcomitans. Las placas fueron incubadas en jarra con vela (para generar atmósfera rica en anhídrido carbónico) durante 7 días a 37ºC. Por otro lado, se tomaron 100 µl de la muestra original de RTF y se obtuvieron diluciones 1:10, 1:100 y 1:1000 en RTF. De éstas, se sembraron 100 µl de las dos últimas, en medio agar base con sangre con menadiona y hemina, incubándose durante 14 días a 37ºC en anaerobiosis estricta para la recuperación de anaerobios (Porphyromonas y Prevotella).
De la muestra en RTF se tomaron muestras de 100 µl además de la muestra original y se almacenaron a -30ºC para luego ser procesadas con la técnica de PCR. Se utilizaron las cepas silvestres como controles positivos: Aggregatibacter actinomycetemcomitans (ATCC 29522), Porphyromonas gingivalis (BAA-308), Prevotella intermedia (ATCC 25611), Tannerella forsythia (ATCC 43037) y Fusobacterium nucleatum (ATCC 25586).
Identificación por cultivo
Las colonias presuntivas de A. actinomycetemcomitans se identificaron por la presencia de una estructura semejante a una estrella en el interior de las colonias con una lupa estereoscópica (Fig. 1), técnica de coloración de Gram, prueba de catalasa (+) y MUG negativa (4-Metilumbeliferil-β-D-galactósido) para investigar la fermentación de la lactosa.
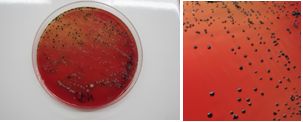
Las bacterias anaerobias pigmentadas (Porphyromonas y Prevotella) se identificaron por la formación de pigmento (Fig. 2). Para su diferenciación, se observaron también mediante fluorescencia bajo luz ultravioleta (360 nm) donde las colonias de P. gingivalis no fluorescen y las de P. intermedia si lo hacen (Fig. 3). En caso de dudas se realizó la identificación con pruebas bioquímicas para anaerobios a través del kit API®.
Colonias fluorescentes de Prevotella intermedia frente a luz ultravioleta
Como forma de validar el estudio se tomó un grupo de control consistente en 10 pacientes seleccionados que no presentaban el cuadro de periodontitis. Como resultado no se obtuvo crecimiento en 8 de los 10 casos en el medio selectivo para A. actinomycetemcomitans. En los dos restantes hubo crecimiento pero no se trataba de A. actinomycetemcomitans. Para el caso de pigmentados se obtuvo crecimiento en 4 de los 10 casos pero con un recuento bajo.
Identificación por PCR. Extracción del ADN genómico microbiano.
Las muestras se descongelaron y se homogenizaron durante 30 segundos con riguroso vórtex. Posteriormente se transfirió el contenido a otro tubo Eppendorf y se centrifugaron a 13500 rpm durante 3 minutos. Se descartó el sobrenadante y nuevamente se suspendió el pellet por pipeteo en 500 µl de RTF durante 5 minutos a 100ºC. Posteriormente se colocaron en hielo por 5 minutos y posterior centrifugación a 13500 rpm por 5 minutos a 4ºC. Finalmente se tomó el sobrenadante conteniendo el ADN genómico microbiano y se almacenó a -30ºC hasta el momento de realizar el PCR.
La extracción del genoma de cada cepa silvestre control, se realizó utilizando el Kit ZymoBeadTMGenomic DNA (como lo describe el fabricante). La determinación de la concentración del ADN (ng/µl) se cuantificó por NanoDrop 2000.
Para el diseño de los cebadores se utilizó la base de datos NCBI (National Center of Biotechnology Information) con la finalidad de buscar y seleccionar las secuencias del 16S ARNr, como blanco en el diseño de los cebadores específicos para T. forsythia (GenBank: AP013045.1) y F. nucleatum (GenBank: MH078456.1). Asimismo, se utilizó información bibliográfica donde se realizaron estudios con oligonucleótidos validados para la especie 19.
Se realizaron PCR con cada par de oligonucleótidos utilizando los genomas extraídos de la cepa silvestre. Los cebadores utilizados para la detección de las especies bacterianas A. actinomycetemcomitans, P. gingivalis, P. intermedia, T. forsythia y F. nucleatum fueron los que se muestran en la Tabla 1. Las condiciones de las PCR multiplex para la detección de estos microorganismos se muestran en las Tablas 2 y 3.
Tabla 1: Diseño de cebadores para la detección de las especies bacterianas presentes en las muestras periodontales
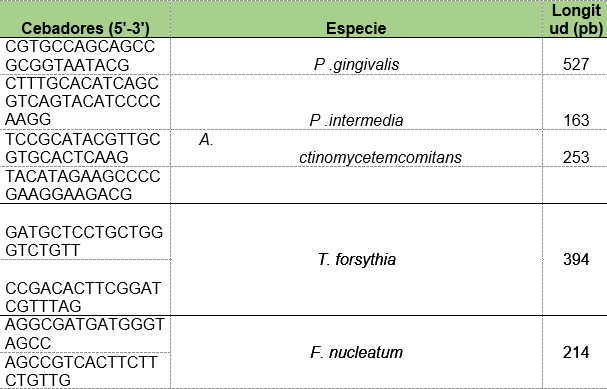
Tabla 3: Condiciones de la PCR multiplex para la detección de P. gingivalis, P. intermedia, A. actinomycetemcomitans

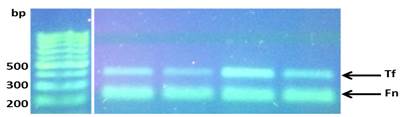
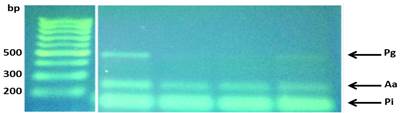
La visualización de los resultados de las amplificaciones (bandas) se realizaron en geles de agarosa al 1.2%, buffer TBE (Tris-borato-EDTA) 0.5x, 5µl de GoodView, 2µl de cyan/orangeloading buffer; 2-5 µl del producto del PCR durante 2h a 80V; y fotografiados con una cámara digital colocada encima de un transiluminador con luz ultravioleta (UV) (Figs. 4 y 5).
Análisis estadístico
Se utilizó estadística descriptiva utilizando frecuencias absolutas en porcentaje. Las comparaciones entre poblaciones se realizaron utilizando Test Student-T con un nivel de significancia de 0.05.
Resultados
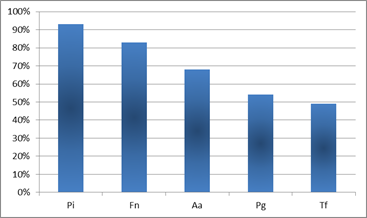
Los microorganismos que aparecieron con mayor prevalencia en los cuadros de Periodontitis Agresiva fueron P. intermedia y F. nucleatum. Éstos aparecieron en el 93% y 83% de los pacientes respectivamente. Le siguió en incidencia A. actinomycetemcomitans, microorganismo que se correlaciona fuertemente con los cuadros de Periodontitis Agresiva 9,11-12,16. A.actinomycetemcomitans apareció en el entorno del 70% de los pacientes estudiados (Graf. 1).
Cuando se hace la distinción entre Periodontitis Agresiva Localizada y Generalizada, la microbiota se diferencia poniendo en relieve la distinta etiopatogenia microbiológica de ambos cuadros. Cabe resaltar, que de los cuadros agresivos estudiados, el 60% de los casos correspondió a casos localizados y el 40% a cuadros generalizados. Es por este motivo que al estudiar la microbiota de los cuadros agresivos sin distinguir entre localizados y generalizados, los porcentajes de prevalencia están condicionados por el mayor número de casos localizados.
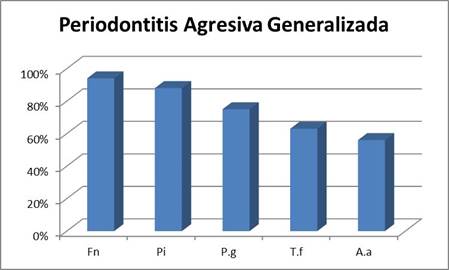
Periodontitis Agresiva Generalizada
Si bien en ambos tipos de cuadros F. nucleatum fue un microorganismo de alta prevalencia, se destacó fundamentalmente en los cuadros agresivos generalizados presentándose en casi el 95% de los pacientes.
En los cuadros generalizados, el microorganismo que le siguió en prevalencia fue P. intermedia (en el entorno del 90%), seguido por P. gingivalis en el 75% de las muestras y por T. forsythia en un 63%. Por otra parte, A. actinomycetemcomitans si bien es recuperado en varios pacientes, fue de los microorganismos estudiados, el menos prevalente presentándose en un 56% (Graf. 2).
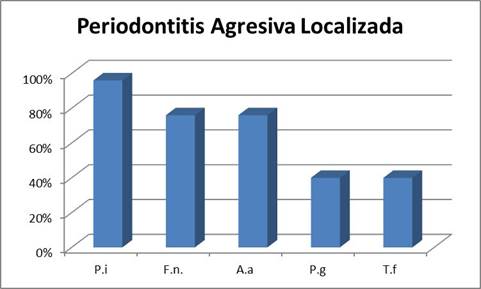
Periodontitis Agresiva Localizada
En cuanto al cuadro localizado el microorganismo más prevalente fue P. intermedia presentándose en el 96% de los pacientes, seguido de F. nucleatum y A. actinomycetemcomitans ambos con una prevalencia en el entorno del 75%. Se destaca la mayor prevalencia de A. actinomycetemcomitans que en los cuadros generalizados. Esto es concordante con la importancia que tiene este microorganismo en los cuadros localizados 13-15. Finalmente, los microorganismos P. gingivalis y T. forsyhtia presentaron la menor prevalencia detectándose en el 40% de los pacientes estudiados (Gráf. 3).
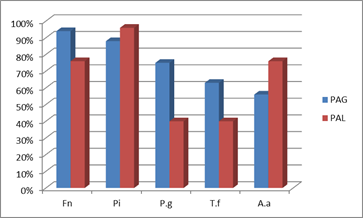
Comparativo de la prevalencia microbiana entre PAG y PAL
Para facilitar la comparación, a continuación se presenta en forma consolidada las respectivas prevalencias de los diferentes patógenos en los cuadros generalizados y localizados en nuestros estudios con pacientes uruguayos (Graf. 4).
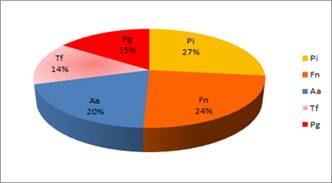
Proporción de los patógenos estudiados en los cuadros de Periodontitis Agresiva
Otra forma de analizar la importancia del microorganismo en el cuadro es estudiándolo en relación a la detección del resto de los patógenos estudiados. Si consideramos como una ocurrencia la detección de un determinado patógeno en un paciente, el 100% de ocurrencias estaría representado por el total de detecciones de los cinco patógenos estudiados. Al estudiar la proporción en que se presenta cada microorganismo en relación al resto de los microrganismos (al total de ocurrencias) se observan las siguientes proporciones (Graf. 5).
El microorganismo que apareció en mayor proporción fue P. intermedia, seguido de F. nucleatum. El tercer lugar correspondió a A. actinomycetemcomitans representando el 20%.
Aquí nuevamente los porcentajes están mayormente influenciados por el mayor número de casos localizados que generalizados.
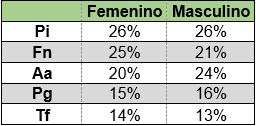
No se detectaron diferencias significativas en la proporción de microorganismos al considerar el sexo con un nivel de significación de 0.05 utilizando la prueba T para dos muestras suponiendo varianzas iguales (Tabla 4).
Discusión
Los microorganismos presentes en la Periodontitis Agresiva Generalizada (PAG) en nuestro estudio son similares a los encontrados en los cuadros de Periodontitis Agresiva en otros países 15-16. De acuerdo a la bibliografía, los microorganismos que suelen detectarse en los cuadros de PAG son P. gingivalis, A. actinomycetemcomitans y T. forsythia16.
En particular se destaca P. gingivalis por sobre A. actinomycetemcomitans en los cuadros generalizados 15.
Respecto a la presencia de los patógenos estudiados en nuestro estudio, se revela que en los cuadros de Periodontitis Agresiva Generalizada sobresalió P. gingivalis sobre A. actinomycetemcomitans en concordancia con la bibliografía y otras localizaciones como Chile y Colombia 20-21.
En cuanto a los cuadros de Periodontitis Agresiva Localizada, en nuestros resultados, A. actinomycetemcomitans superó a los periodontopatógenos P. gingivalis y T. forsythia similarmente a otras localizaciones como en Europa y Norteamérica 2,11,15,21 donde se recupera A. actinomycetemcomitans especialmente en los cuadros localizados. Sin embargo, en estudios en Latinoamérica (Chile, Colombia) la recuperación de P. gingivalis y T. forsythia superó a A. actinomycetemcomitans en estos cuadros 20,22-23.
Es interesante también mencionar que algunos autores remarcan la importancia de A. actinomycetemcomitans en su rol en la patogénesis de la Periodontitis Agresiva, en especial en la forma localizada, si bien se ha planteado que es probable su particular importancia en el comienzo de la patología para luego ser reemplazada por otras bacterias patógenas a medida que progresa la enfermedad 11,13.
Conclusiones
Se verificó una flora diferenciada entre los cuadros localizados y los generalizados en los pacientes uruguayos estudiados.
El microorganismo más prevalente de la Periodontitis Agresiva Localizada fue P. intermedia mientras que en la Periodontitis Agresiva Generalizada fue F. nucleatum.
Por otro lado, en los cuadros generalizados los microorganismos P. gingivalis y T. forshytia son más prevalentes que en los cuadros localizados.
Asimismo, confirmamos la importancia del microorganismo A. actinomycetemcomitans en los cuadros de Periodontitis Agresiva, destacándose en los cuadros localizados.
En nuestro país se presentó una flora similar en los cuadros agresivos a los que figuran en otras localizaciones geográficas, si bien en algunas de éstas se destaca P. gingivalis sobre A. actinomycetemcomitans en todos los cuadros de Periodontitis Agresiva (20, 22). Esta relación, en nuestro caso, se da sólo en los cuadros agresivos generalizados. Por otro lado, en los cuadros agresivos localizados el microorganismo A. actinomycetemcomitans apareció en mayor proporción a P. gingivalis lo cual es concordante con los datos reportados en la bibliografía 16.
Se pudo además correlacionar el estudio bacteriológico tradicional con técnicas de estudio molecular.










 texto en
texto en