Introducción
La Hepatozoonosis es una parasitosis causada por protozoarios apicomplexos de la familia Hepatozoidae, pertenecientes al género Hepatozoon. En la actualidad, se han descrito más de 340 especies de Hepatozoon en vertebrados (anfibios, reptiles, aves y mamíferos), de las cuales, unas 50 han sido reportadas en mamíferos domésticos y salvajes (Baneth et al., 2013; Smith, 1996). Estudios recientes han sugerido que Hepatozoon puede comprender a más de un género, ya que contiene diferentes linajes (Karadjian et al., 2015; Kvičerová et al., 2014); sin embargo, la división del género se considera controvertida (Maia et al., 2016).
El ciclo de Hepatozoon spp. incluye una forma de desarrollo asexual llamada merogonia, seguido de gametogonia en el vertebrado intermediario, que puede ser un mamífero, y un desarrollo sexual que lleva a la esporogonia en el invertebrado hematófago, que es el hospedador definitivo (Smith, 1996). Los principales hospedadores definitivos invertebrados de Hepatozoon spp. son una amplia variedad de artrópodos hematófagos como, garrapatas (Ixodidae y Argasidae), ácaros (Acari), mosquitos (Culicidae), flebótomos (Phlebotominae), moscas picadoras (Diptera), pulgas (Siphonaptera), piojos (Anoplura) y chinches (Hemiptera). La ingestión de estos invertebrados (hospedadores definitivos) que contienen ooquistes maduros, por los hospedadores vertebrados (hospedadores intermediarios), es una de las principales vías de infección, aunque también se ha reportado la transmisión transplacentaria y por depredación (Baneth et al., 2013; Johnson et al., 2009; Murata et al., 1993). Se ha observado en varias especies de Hepatozoon la presencia de pequeños quistes tisulares que contienen etapas parasitarias únicas o múltiples (cistozoitos), estos quistes tisulares están asociados con la transmisión por depredación (Baneth y Shkap, 2003; Baneth et al., 2007; Paperna et al., 2002; Smith et al., 1994; Vincent-Johnson et al., 1997). Esto resalta la complejidad de los ciclos biológicos en las diferentes especies de este género (Smith, 1996).
Varias especies de Hepatozoon han sido reportadas parasitando carnívoros domésticos y silvestres. El perro doméstico es parasitado por dos especies, Hepatozoon americanum y Hepatozoon canis. En el caso de H. americanum es transmitido por la garrapata Amblyomma maculatum y afecta perros en el sureste y centro-sur de Estados Unidos de América (EE.UU.). En cambio, para H. canis el vector primario es Rhipicephalus sanguineus, aunque otras garrapatas también pueden estar involucradas, y posee una amplia distribución mundial. Hepatozoon canis y H. americanum pueden ocasionar cuadros clínicos que pueden ir desde asintomáticos hasta provocar la muerte de los perros (Little et al., 2009; Vincent-Johnson, 2014). En el gato doméstico, el primer reporte de Hepatozoon corresponde a Patton (1908), quien lo detectó en sangre de un gato en la India, y lo denominó inicialmente como Leucocytozoon felis domestici. Luego, fue transferido al género Hepatozoon, combinándose como Hepatozoon felis. Asimismo, debido a la similitud en la morfología de los gamontes observados en sangre, se sugirió que los parásitos de este género hallados en gato, chacal y hiena eran indistinguibles del H. canis que infecta a los perros (Wenyon, 1926). En la actualidad, H. felis fue redescrito e identificado como una especie diferente (Baneth et al., 2013).
Actualmente, se conocen tres especies de Hepatozoon que parasitan felinos, de las cuales H. felis es considerada la especie más prevalente, H. canis es de hallazgo esporádico, y recientemente, Hepatozoon silvestris fue reportada parasitando al gato silvestre europeo (Felis silvestris silvestris) en Bosnia y Herzegovina (Baneth et al., 2013; Díaz-Regañón et al., 2017; Hodžić et al., 2016; Jittapalapong et al., 2006).
Cuando la hepatozoonosis en gatos es causada por H. felis, es generalmente subclínica, ya que una alta proporción de la población felina está infectada sin manifestaciones clínicas (Baneth et al., 2013). La hepatozoonosis clínica en gatos se ha reportado cuando el mismo presenta una co-infección con enfermedades inmunodepresoras, como ser, el virus de la leucemia felina (FeLV), el virus de la inmunodeficiencia felina (FIV), micoplasmas o leishmaniosis (Lloret et al., 2015; Vincent-Johnson, 2014). En estos casos se han observado un amplio rango de signos clínicos, entre ellos debilidad, anorexia, pérdida de peso, fiebre, linfadenomegalia, glositis ulcerativa, hipersalivación, anemia, secreción ocular serosa e ictericia y anormalidades clinicopatológicas como anemia y trombocitopenia (Lloret et al., 2015).
El objetivo del presente trabajo es dar a conocer la detección mediante técnicas moleculares de Hepatozoon felis en gatos que presentaban anemia clínica en Uruguay.
Materiales y métodos
Obtención de las muestras
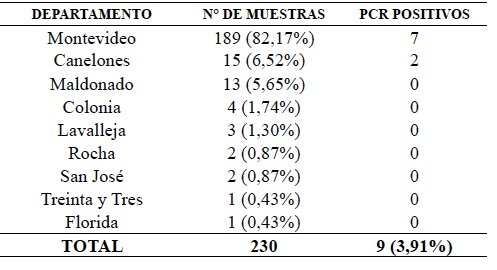
Para este estudio se obtuvieron 230 muestras de sangre con anticoagulante de gatos domésticos a los cuales previamente se determinó la presencia de anemia mediante hemograma en el Laboratorio Clínico Veterinario (Laclivet-Montevideo). Las muestras fueron colectadas entre noviembre de 2015 y diciembre de 2019, las cuales procedieron de nueve departamentos de Uruguay (Tabla 1). Las mismas fueron enviadas al Laboratorio de Vectores y Enfermedades Transmitidas (LVyET), CENUR Litoral Norte-Salto, UdelaR, para realizar el diagnóstico molecular de hemoparásitos.
Estudios paraclínicos
Además del hemograma completo a través del cual se determinó anemia, se realizaron, en algunos casos, estudios sobre funcionalidad hepática (GOT/AST, GPT/ALT, Fosfatasa Alcalina, Bilirrubina total, directa e indirecta) y renal (urea, creatinina).
Extracción de ADN, amplificación (PCR) y secuenciación
Para los estudios moleculares, la extracción de ADN se llevó a cabo a partir de 200 µl de sangre entera utilizando el kit comercial GeneJET Genomic DNA Purification Kit (Thermo Fisher Scientific), siguiendo las instrucciones del fabricante. El ADN obtenido fue cuantificado y evaluada su pureza por espectrofotometría mediante el uso de un equipo NanoDrop 2000 (Thermo Fisher Scientific, Waltham, MA, USA). Para la detección de ADN de Hepatozoon spp. se realizaron dos PCRs independientes utilizando los cebadores HEMO1 (5ˊ-TAT TGG TTT TAA GAA CTA ATT TTA TGA TTG-3´) y HEMO2 (5´-CTT CTC CTT CCT TTA AGT GAT AAG GTT CAC-3´) que amplifican un fragmento de ≈900 pb del gen 18S ARN ribosomal (rARN) (Perkins y Keller, 2001), y los cebadores HEP1mod (5´-CGC GAA ATT ACC CAA TTC TA - 3´) y HEP4 (5´-TAA GGT GCT GAA GGA GTC GTT TAT-3´) que amplifican un fragmento de ≈670 pb del mismo gen (Spolidorio et al., 2009). Las condiciones de PCR para los cebadores HEMO son descritas por Harris et al. (2011), y para los cebadores HEP por Spolidorio et al. (2009). Se amplificaron fragmentos solapantes, a fin de obtener una secuencia de ≈1.300 pb. Se utilizó ADN de Hepatozoon sp. obtenido de una culebra parejera (Philodryas patagoniensis) como control positivo y agua libre de ADN como control negativo en cada reacción. Los productos resultantes de las PCRs se verificaron mediante electroforesis en gel de agarosa al 1.5%, que contenía el intercalante GoodViewTM Nucleic Acid Stain (Beijing SBS Genetech Co., Ltd.), y examinados mediante un transiluminador UV. Los amplicones del tamaño esperado se purificaron mediante el kit comercial GeneJET PCR Purification Kit (Thermo Fisher Scientific) y se enviaron para secuenciar a la empresa MACROGEN (Seúl, Corea del Sur).
Análisis filogenéticos
Las secuencias obtenidas se corrigieron manualmente y se alinearon utilizando el programa MUSCLE del software MEGA 7 (Kumar et al., 2016). Las secuencias consenso se lograron combinando las secuencias de los dos fragmentos parcialmente solapantes del gen 18S rARN. Para la determinación de la identidad, las secuencias obtenidas se compararon con secuencias disponibles en el GenBank mediante BLAST® http://www.ncbi.nlm.nih.gov/BLAST). Para el análisis filogenético se realizó un alineamiento utilizando las secuencias obtenidas en este estudio, junto a otras descargadas de GenBank y se construyó un árbol filogenético utilizando el método de Máxima Verosimilitud, a través del modelo Tamura 3 parámetros (Tamura, 1992). El soporte de los nodos fue determinado usando 1000 réplicas de Bootstrap. Como grupo externo se utilizó Adelina grylli (acceso GenBank: DQ096836).
Resultados
Diagnóstico mediante PCR y secuenciación
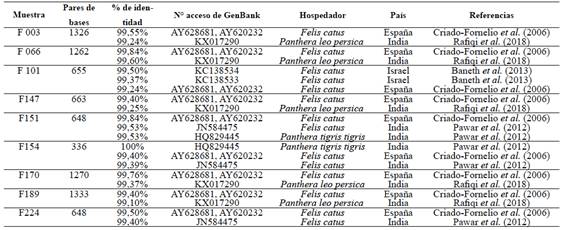
De las 230 muestras de sangre de gatos analizadas, nueve amplificaron para las PCRs realizadas. Por lo que la frecuencia de infección fue de 3,91% (9/230). Si bien se visualizaron bandas del tamaño esperado para cada PCR, luego de la secuenciación, la calidad de las secuencias obtenidas fue variada. Por lo tanto, en algunas muestras, las secuencias consenso obtenidas fueron más cortas. A través del análisis en BLAST®, todas las secuencias demostraron tener una identidad entre 99,10 y 100% con secuencias de H. felis depositadas en Genbank. El largo de cada secuencia obtenida y el porcentaje de identidad se detallan en la Tabla 2. Las secuencias de las muestras F003, F066, F101, F151, F170, F189 que fueron utilizadas en el análisis filogenético, fueron depositadas en el GenBank bajo los números de acceso: MT210593, MT210594, MT210595, MT210596, MT210597 y MT210598, respectivamente.
Análisis filogenético
Los resultados de la construcción del árbol filogenético indican que las secuencias de este estudio se agrupan en un clado con un alto soporte (Bootstrap=97) con secuencias correspondientes a H. felis provenientes de diferentes continentes (Figura 1).
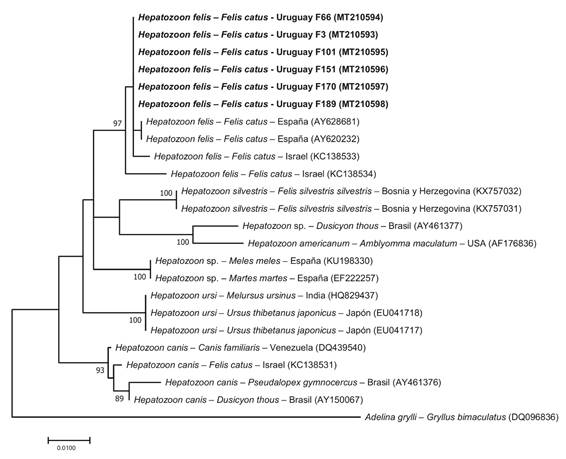
Figura 1: Árbol filogenético construido con el método de máxima verosimilitud utilizando secuencias del gen 18S rARN de Hepatozoon spp. Se utilizaron 24 secuencias, Adelina grylli se incluyó como grupo externo. En el nombre de las secuencias se detalla especie, hospedero y números de acceso de GenBank. Se resaltan las secuencias obtenidas en este estudio en negrita y se muestran los valores de soporte de rama mayores a 70.
Estudios paraclínicos
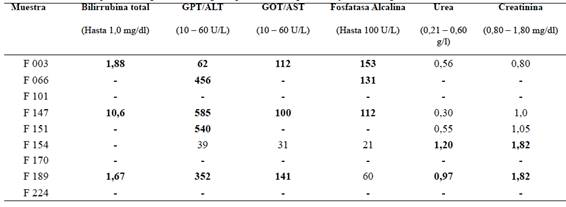
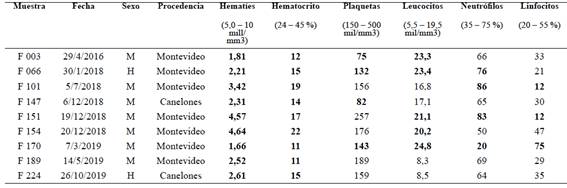
Los resultados de hemograma, funcional hepático y renal de los gatos positivos a H. felis se muestran en las Tablas 3 y 4, respectivamente. Como ya fue mencionado, todas las muestras de sangre procesadas provinieron de gatos con diagnóstico de anemia. Otras alteraciones citopatológicas observadas fueron trombocitopenia (n=4), hiperleucocitosis (n=5) y neutrofilia (n=4). Tres de los cinco gatos que les realizaron estudios de funcionalidad hepática y renal presentaron bilirrubina total aumentada. Adicionalmente, los perfiles enzimáticos de GPT/ALT se observaron sumamente marcados en cuatro de seis gatos y de GOT/AST en tres de cuatro. La fosfatasa alcalina se halló elevada en tres de cinco gatos. Urea y creatinina se hallaron aumentadas en dos de cinco gatos.
Tabla 3: Datos de hemograma de los gatos positivos a Hepatozoom felis. En negrita se resaltan los valores alterados.
Discusión
La hepatozoonosis felina por H. felis es, en su mayoría, una afección subclínica ya que una gran parte de la población está infectada sin manifestaciones clínicas. Por lo que la parasitosis se desarrollaría en asociación con una inmunodeficiencia inducida por una enfermedad concomitante (Baneth et al., 1998, 2013). Los estudios moleculares llevados a cabo en este trabajo indicaron que todas las muestras positivas a Hepatozoon correspondieron a H. felis, con una frecuencia de infección de 3.91%. En otro estudio llevado a cabo mediante técnicas moleculares en España, Criado-Fornelio et al. (2006) hallaron únicamente dos gatos positivos a H. felis de 330 estudiados (0,6%). En cambio, Baneth et al. (2013) estudiaron en Israel 152 gatos, de los cuales, 55 fueron positivos a Hepatozoon sp. (36,2%). Las diferencias en las frecuencias de infección del presente trabajo y los anteriores podrían ser explicadas por la diferencia en los hábitos de los gatos estudiados. En el estudio de Baneth et al. (2013), casi la mitad de los gatos que estaban al aire libre o pasaban gran parte del día fuera de su hogar fueron positivos a Hepatozoon sp., mientras que solo el 10% de los que permanecían estrictamente en el interior, resultaron positivos. Esto demuestra una significativa asociación entre los animales positivos a Hepatozoon sp. y el acceso al exterior. Esta asociación también fue observada en el estudio de Jittapalapong et al. (2006) en Tailandia, donde de 300 gatos “callejeros” analizados, el 32,3% resultaron positivos a Hepatozoon sp. Esta relación entre la alta frecuencia de infección por Hepatozoon sp. y el acceso al exterior de los gatos podría deberse a una mayor disponibilidad a hospedadores definitivos como garrapatas, pulgas o ácaros, y/o también mayor posibilidad de predación. Si bien en nuestro estudio no poseemos información sobre los hábitos de los gatos estudiados, la mayoría fueron remitidos por médicos veterinarios del departamento de Montevideo (189/230, 82,17%), por lo tanto, es probable que la mayoría de ellos tengan escaso acceso al exterior, así como cierto control de ectoparásitos que puedan actuar como fuente de infección. En concordancia con Baneth et al. (1998), es posible que la mayoría de las anomalías hematológicas, hepáticas y renales halladas en los análisis paraclínicos realizados en los gatos positivos a H. felis, se puedan atribuir a la presencia de otras patologías probablemente inmunosupresoras en los gatos de este trabajo. En el estudio de Baneth et al. (1998), tres de seis gatos fueron positivos al virus de la inmunodeficiencia felina (VIF) y uno al virus de la leucemia felina (VILEF). Uno de los infectados con VIF y el infectado con VILEF resultaron también positivos a Hemobartonella felis. Estos felinos comparten alteraciones como anemia, trombocitopenia, hiperleucocitosis, linfopenia y neutrofilia, con los gatos positivos a H. felis de este trabajo. Si bien no podemos relacionar de manera directa estas alteraciones, ya que no se contó con un diagnóstico previo de otras infecciones concurrentes, es altamente probable que otro proceso infeccioso mayor haya sido el causante de las alteraciones reportadas y no debidas a una hepatozoonosis como parasitosis o patología única.
La mayoría de los gatos positivos a H. felis en este estudio fueron machos (7/9) (Tabla 3), lo cual se puede explicar debido a su comportamiento diferencial frente al de las hembras. Los machos son territoriales y tienden a pelear más que las hembras, por lo tanto, son más propensos a infectarse con virus inmunosupresores como el VIF y el VILEF, pudiendo volverlos aún más susceptibles a la infección por Hepatozoon spp. (Baneth et al., 1998). En los felinos que se manifiesta la enfermedad, Hepatozoon sp. podría ser un parásito oportunista que infecta gatos inmunosuprimidos, o podría ya estar presente como una infección latente en animales clínicamente sanos y que la enfermedad se manifieste en condiciones de estrés o de inmunosupresión (Baneth et al., 1998).
Debido a problemas en la calidad de las secuencias, algunas de las mismas resultaron ser más cortas. Por lo tanto, no pudieron ser incluidas en el árbol filogenético. El alineamiento constó de 23 secuencias de diferentes especies de Hepatozoon. La descripción del análisis filogenético basada en la comparación de las secuencias nucleotídicas del gen 18S rARN de diferentes especies de Hepatozoon indicó que las secuencias generadas en este trabajo se agruparon en un clado con alto soporte de Bootstrap (97) con secuencias de H. felis descritas parasitando gatos de España e Israel correspondiendo a la misma especie (Figura 1). En concordancia con Baneth et al., (2013) nuestros hallazgos demuestran que H. felis descrito en este trabajo se distingue claramente de H. canis y de las otras especies de Hepatozoon utilizadas para el análisis filogenético.