Introducción
El género Staphylococcus incluye una amplia variedad de especies bacterianas que se encuentran ampliamente distribuidas en la naturaleza colonizando mamíferos y aves. Se encuentran normalmente formando parte de la microbiota natural de la piel, del tracto respiratorio, del tracto urogenital y transitoriamente del tracto digestivo tanto en animales como en el hombre.
Son cocos Gram positivos anaerobios facultativos, halófilos, catalasa positivos, inmóviles, no esporulados, con un metabolismo fermentativo y oxidasa negativos, además, son ampliamente resistentes a la sequedad y la desinfección (Bannoehr y Guardabassi, 2012).
Este género bacteriano presenta 45 especies y 21 subespecies que se clasifican dentro de dos grupos teniendo en cuenta su capacidad de producir la enzima coagulasa: estafilococos coagulasa negativos (CoNS) y estafilococos coagulasa positivos (CoPS) pudiendo ser ambos potenciales patógenos zoonóticos de interés en medicina humana y veterinaria. (Bergeron et al., 2011; Couto et al., 2016; de Godoy et al., 2016).
La proliferación y diseminación al medio de estos microorganismos resistentes a antibióticos es uno de los problemas actuales de mayor relevancia tanto en medicina humana como veterinaria.
En el año 1972 se reportó el primer aislamiento de Staphylococcus aureus resistente a meticilina (MRSA) en animales a partir de muestras de leche de vacas que tenían mastitis (Devriese et al., 1972). Al igual que pasó en humanos, desde este primer caso hasta el presente se han reportado un importante número de cepas de MRSA en una gran variedad de especies domésticas como perros, gatos, cerdos, caballos, ovejas y aves (Leonard y Markey, 2008).
Como el MRSA los aislamientos de cepas de S. pseudintermedius meticilino resistente (MRSP) en perros también han aumentado drásticamente, mostrando resistencia a una gran variedad de antibióticos utilizados comúnmente en medicina veterinaria y siendo considerados grandes reservorios de genes de resistencia a antimicrobianos (Perreten et al., 2013).
Por otro lado, los aislamientos de CoNS como responsables de infecciones ha ido en aumento llevando a un mayor interés en el estudio de sus mecanismos de resistencia por considerarlos un gran reservorio de estos genes y con un gran potencial de transferirlos a otras especies de estafilococos (Czekaj et al., 2015; Piette y Verschraegen, 2009).
Por ejemplo, el S. haemolyticus resistente a meticilina ha sido reportado tanto en aislamientos de humanos (Barros et al., 2012) como de perros, así como también, se han reportado una gran cantidad de cepas de S. epidermidis resistentes a meticilina que están ampliamente diseminadas (Piette y Verschraegen, 2009; Vanderhaeghen et al. 2012).
La resistencia a meticilina (y oxacilina) por parte de los Staphylococcus sp. es debida a la presencia de una proteína PBP de baja afinidad por los β-lactámicos, denominada PBP 2a o PBP 2´ para diferenciarla de la PBP nativa. El gen que codifica para esta proteína es el mecA que está presente en la mayoría de las cepas de estafilococos resistentes estando muy conservado en estas cepas y localizado dentro de un elemento genético móvil llamado SCCmec (Staphylococcal cassette chromosomal) (Gradelski et al., 2001; Mann P, 1959).
Hasta el momento no se han realizado estudios de este tipo en caninos en el Uruguay, lo cual es llamativo dado el rol de las enfermedades que ellos producen y los antecedentes internacionales que relacionan al contagio entre animales y humanos. Creemos que existe una amplia circulación de diferentes especies de Staphylococcus mayoritariamente aquellas aisladas comúnmente en perros (S. xylosus, S. pseudintermedius, S. epidermidis, S. haemolyticus) las cuales tienen una frecuencia de resistencia a meticilina comparable a los datos obtenidos en la región y el mundo.
El objetivo de este trabajo fue detectar cepas de Staphylococcus sp. resistentes a meticilina en perros sanos y con sintomatología característica (piodermia y/o otitis) muestreados en Montevideo durante un período de un año (mayo de 2016 - mayo de 2017), así como también determinar su perfil de resistencia frente a una serie de antibióticos.
Materiales y métodos
Recolección de muestras
El muestreo (n=55 perros) fue realizado desde mayo de 2016 a mayo de 2017 por profesionales veterinarios que desempeñaban funciones en el Centro Hospitalario de la Facultad de Veterinaria de la Universidad de la República, Montevideo, Uruguay. De los perros muestreados 25 fueron perros enfermos que ingresaron al hospital por problemas de piel u oído mientras que 30 perros fueron categorizados como sanos y fueron ingresados al hospital por otras dolencias no relacionadas a la infección por Staphylococcus (afecciones traumatológicas, nerviosas, renales, cardíacas etc). Se analizó un total de 67 aislamientos, 30 faríngeos, 12 perianales, 23 de lesiones de piel y 2 de oído. Los perros muestreados fueron cachorros (de menos de un año), adultos (1 a 7 años) y gerontes (mayores de 7 años), todos sin tratamiento con antibióticos.
Caracterización fenotípica de los aislamientos
Los hisopos se cultivaron en medio líquido de enriquecimiento Tryptic Soy Broth (TSB) 10% NaCl (Oxoid) y se incubaron a 37°C durante 18 ± 2hs. Posteriormente a partir de los caldos crecidos se realizó el aislamiento en Manitol Sal Agar (MSA) (Oxoid) y las placas se incubaron 18 ± 2hs a 37°C. Todas las placas de MSA con crecimiento se reaislaron en placas de Tripticasa Soy Agar (TSA) y se les realizó tinción de Gram, catalasa, coagulasa (BBL Coagulase Plasma, Rabbit con EDTA®), Voges Proskauer y presencia de β-galactosidasa (sembrando en medio TSA con X-Gal) (Tse et al., 2012).
Extracción del ADN genómico
La extracción del ADN genómico se realizó utilizando la enzima achromopeptidasa (Sigma-Aldrich® St. Louis, MO). Trabajando en cabina de flujo laminar se colocaron 95 μl de agua ultrapura estéril en tubos para microcentrífuga de 1,5 ml estériles. Posteriormente se le agregaron 5 µl de la solución de achromopeptidasa a una concentración de 10U/µl y se resuspendieron 2 o 3 colonias en el tubo.
Los tubos se colocaron en un baño a 50°C durante 10 minutos seguido de un baño a 94°C durante 10 minutos más. Pasado este tiempo se centrifugaron a 13.500 rpm durante 5 minutos, 80-90 µl del sobrenadante se transfirieron a un tubo nuevo, se evaluó su pureza mediante NanoDrop y se guardó a -20°C hasta su uso en la PCR.
Identificación de los aislamientos por amplificación y secuenciación del ARNr 16S
La identidad de todos los aislamientos se confirmó mediante la amplificación y secuenciación del ARNr 16S. Para la amplificación de este gen se utilizaron dos pares de primers cuyos fragmento de amplificación se superponen en 124 nucleótidos, según su posición en la secuencia de referencia de E. coli(Martínez-Rosales y Castro-Sowinski, 2011). Cada muestra se amplificó con los dos pares de primers para luego reconstruir la secuencia completa. Las reacciones de PCR se realizaron utilizando MangoMix™ de Bioline y la mezcla para las reacciones de PCR se realizaron en un volumen final de 25 µL: 12,5 µL de MangoMix™, 1 µL de cada primer a una concentración de 10 µM, 9,5 µL de H2O mQ estéril y de 50-100 ng de ADN. La detección de los amplicones se realizó mediante corrida electroforética a 90V en un gel de agarosa al 1% conteniendo el intercalante GoodView® Nucleic Acid Stain (Beijing SBS Genetech Co., LTd) que se visualizó utilizando un transiluminador UV.
Una vez obtenidas las secuencias de cada amplicón se empalmaron y editaron mediante el programa Bioedit para obtener un fragmento de aproximadamente 1400 pb. La identificación de las cepas se realizó utilizando el servidor online www.ezbiocloud.net. desarrollado y mantenido por el laboratorio “ChunLab” (http://chunlab.com) (Yoon et al., 2017).
Test de susceptibilidad a antibióticos
Los análisis de susceptibilidad se realizaron por medio de antibiogramas siguiendo el método de Disco difusión de Kirby - Bauer bajo las recomendaciones del Clinical & Laboratory Standards Institute (CLSI VET01S. 3th ed y CLSI M100. 27th ed). Se analizó la susceptibilidad a 10 antibióticos (Rubin et al., 2011) utilizando como controles positivos de resistencia a meticilina una cepa de S. aureus meticilino resistente (MRSA) y una cepa de S. pseudintermedius meticilino resistente (MRSP) cedidas por el Laboratorio de Bacteriología del Instituto de Higiene de la Universidad de la República del Uruguay, como control negativo se utilizó la cepa de referencia de S. aureus ATCC 6538.
Los antibióticos fueron seleccionados teniendo en cuenta el uso que se les da en medicina veterinaria y también con el objetivo de buscar resistencias nuevas en las cepas aisladas. Se testearon los siguientes antibióticos: oxacilina, penicilina, amoxicilina/clavulánico, ampicilina/sulbactam, cefalotina, enrofloxacina, gentamicina, vancomicina, eritromicina y kanamicina. Se clasificaron como cepas resistententes a múltiples drogas (MDR, del inglés multidrug-resistant) todas aquellas cepas que mostraron resistencia a tres o más clases de antibióticos (Magiorakos et al., 2012).
Detección del gen mecA por PCR
Para la detección del gen mecA por PCR se utilizaron primers previamente publicados (Jonas et al., 2002) y así obtener un tamaño de banda de 310 pb. La mezcla para las reacciones de PCR se realizaron en un volumen final de 25 µL: 12,5 µL de MangoMix™, 1,5 µL de cada primer a una concentración de 10 µM, 9 µL de H2O mQ estéril y 50 - 100 ng de ADN. Como control positivo se utilizaron las cepas de S. aureus y S. pseudintermedius resistentes a meticilina cedidas por el Laboratorio de Bacteriología del Instituto de Higiene de la Universidad de la República y la cepa de S. aureus ATCC 6538 como control negativo. La detección de los amplicones se realizó mediante corrida electroforética en un gel de agarosa al 1,5 % a 100 V. Los productos de PCR fueron purificados y secuenciados por Macrogen Inc., Seúl, Corea del Sur. Para obtener la secuencia, el cromatograma recibido desde el servicio de secuenciación fue editado en el programa Bioedit, mediante inspección visual se eliminaron los errores típicos del secuenciador Sanger. La secuencia de los primers forward (directo) y reverse (reverso) se empalmaron para conseguir una secuencia única también utilizando el programa Bioedit.
Resultados
Identificación fenotípica y por secuenciación del ARNr 16S
Desde el punto de vista fenotípico los 67 aislamientos pudieron ser agrupados dentro del género Staphylococcus sp. y se pudo determinar, mediante la prueba de la coagulasa, que el 52% de los aislamientos eran Staphylococcus coagulasa negativas mientras que el 48% eran Staphylococcus coagulasa positivos.
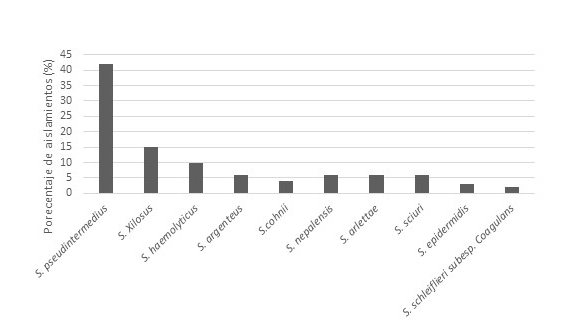
La identificación mediante secuenciación del gen que codifica para el ARNr 16S permitió realizar una identificación más precisa a nivel de especie determinando que la mayoría de los aislamientos obtenidos eran de S. pseudintermedius seguidos por cepas de S. xylosus y S. haemolíticus. Las especies S. argenteus, S. cohnii, S. nepalensis, S. arlettae, S. sciuri, S. epidermidis y S. schleiferi se obtuvieron en menor proporción (Figura 1).
Resistencia a nivel fenotípico y genotípico
La resistencia observada frente a los diferentes antibióticos testados fue bastante variada dentro de las 67 cepas analizadas (Figura 2).
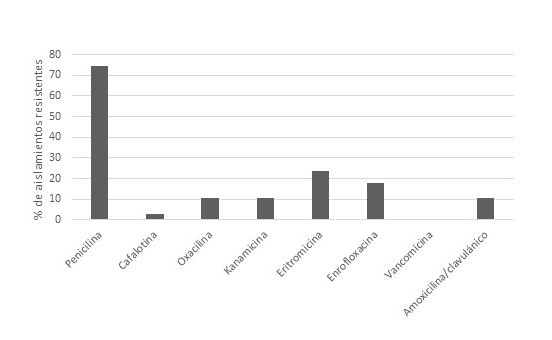
Figura 2: Porcentajes de aislamientos resistentes a cada uno de los antibióticos testeados: penicilina 75%; cefalotina 3%; oxacilina 10%; kanamicina 10%; eritromicina 24%; enrofloxacina 18%; vancomicina 0% y amoxicilna / clavulánico 4%.
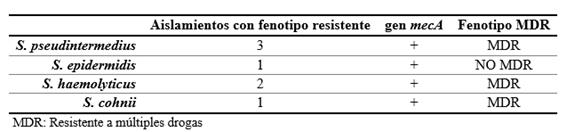
La resistencia a la penicilina se observó en 17 cepas, lo que arrojó un gran porcentaje de cepas resistentes a este antibiótico. La resistencia a cefalotina fue prácticamente nula limitándose a dos cepas de las 67. Con respecto a la vancomicina las 67 cepas analizadas fueron sensibles a este antibiótico y solamente siete cepas fueron resistentes a kanamicina, cuatro de ellas resistentes también a meticilina. 12 cepas mostraron resistencia frente a enrofloxacina y 16 cepas fueron resistentes a eritromicina (Figura 2). Particularmente en cuanto a le resistencia a meticilina, tres cepas S. pseudintermedius, una de S. epidermidis, dos de S. haemolyticus y una de S. cohnii, resultaron resistentes a meticilina. En los antibiogramas de estas cepas no se observó halo de inhibición en el disco de oxacilina y esta resistencia fue confirmada luego por detección por PCR del gen mecA.
Finalmente, todas las cepas que eran meticilino resistentes se consideraron también resistentes a amoxicilina/Clavulánico (Figura 2).
Se obtuvieron 10 cepas que fenotípicamente se podían clasificar como MDR (resistentes a múltiples drogas). Siete de estas diez cepas fueron S. pseudintermedius mientras que se obtuvo un aislamiento de S. chonii, S. epidermidis y S.argenteus MDR. Es importante destacar que tres de estas cepas de S. psudintermedius MDR además eran resistentes a meticilina así como también la cepa de S.epidermidis y S. cohnii MDR (Tabla 1).
Detección del gen mecA por PCR
De las 7 cepas resistentes a meticilina y portadoras del gen mecA 5 fueron obtenidas de faringe de perros sanos mientras que solo dos cepas resistentes a meticilina fueron aisladas de lesiones de piel. En las siete cepas se logró amplificar el gen mecA y se observó una banda de tamaño esperado que correspondía a 310 pb aproximadamente. La secuencia obtenida de los amplicones presentaban alta calidad y una vez editadas todas tenían un 100% de similitud con genes mecA de S. aureus de la base de datos del GenBank.
Discusión
Diversidad de las cepas aisladas y su resistencia a meticilina
En varios trabajos se ha determinado que aproximadamente el 90% de los perros sanos están colonizados por S. pseudintermedius, ya sea a nivel de piel o en mucosas (Bannoehr y Guardabassi, 2012). No es extraño entonces que casi un 50% de los aislamientos que se obtuvieron de los perros muestreados pertenezca a esta especie. Además clínicamente esta especie es la causa más común de piodermia, otitis e infecciones urinarias en los perros (van Duijkeren y Catry, 2011).
El porcentaje de resistencia a meticilina que se obtuvo en este trabajo para la especie S. pseudintermedius (un poco más de 10%) concuerda con lo publicado en el año 2017 por Gronthal en Finlandia (Grönthal et al., 2017) que determinó que el porcentaje de cepas de esta especie resistentes a meticilina era de un 14%, en un total de 1958 aislamientos obtenidos de perros.
Las cepas meticilino resistentes se han relacionado siempre con infecciones y patologías de piel, el aislamiento faríngeo de estas cepas en nuestro trabajo demuestra su importancia en la transmisión al humano ya que es una zona del perro que está en estrecho contacto con las personas (lamidos y mordeduras) y no así las heridas o abscesos de piel.
Todas las cepas de S. xylosus obtenidas fueron aisladas de muestras de faringe por lo que podríamos suponer que esta especie se habría adaptado a colonizar preferentemente este nicho en los perros.
El S. argenteus es una especie similar fenotípicamente a S. aureus pero cuando crecen en medios de cultivo simples las colonias presentan un color blanco y no amarillas debido a que carecen de un carotenoide llamado staphyloxantina (Tong et al., 2013). Trabajos posteriores llegaron a una clasificación taxonómica formal de estas cepas como S. argenteus sp. nov y han sido aislados y asociados a MRSA comunitarios presentes en el Norte de Australia, Camboya, Indonesia, Fiji y Trinidad y Tobago, Europa y África (Jenney et al., 2014; Monecke et al., 2014; Tong et al., 2013). Estos trabajos concluyen que la especie S. argenteus estaría en un proceso de dispersión que ha comenzado recientemente, aumentando su frecuencia de aparición. En animales existe un único reporte, una cepa de S. argenteus fue aislada de materia fecal de un murciélago frugívoro, lo que indicaría que esta especie podría también encontrarse en otras especies de animales (Akobi et al., 2012). El aislamiento correspondiente a S. argenteus de nuestro trabajo confirma esta observación y posiciona a los perros como posible elemento asociado a la dispersión y reservorio. Hasta donde sabemos los aislamientos obtenidos en este trabajo serían los primeros de la especie en perros y tampoco hay reportes de resistencia en esta especie.
Con respecto a las cepas de S. sciuri sabemos que es una especie que puede ser aislada de piel y mucosas de una amplio rango de animales, incluyendo mascotas y animales de granja (Hauschild y Schwarz, 2003). Se aislaron cinco cepas de esta especie en este relevamiento.
En el año 2015, en Dublin, Irlanda, McManus y colaboradores aislaron 40 cepas de S. epidermidis de perros, siendo 33 resistentes a meticilina, esto representa un 82,5% de resistencia a este antibiótico (McManus et al., 2015). De las dos cepas de esta especie que se aislaron en este trabajo una de ellas era resistente a meticilina. Varios estudios han demostrado que la resistencia a meticilina es más común en cepas de S. epidermidis y S. haemolyticum que en las cepas de S. aureus tanto en aislamientos de animales como en humanos (Garza-González et al., 2010), por lo que es posible que sea necesario aumentar el muestreo para aproximarnos al porcentaje real de resistencia en perros de Montevideo.
La especie de S. conhii es, como muchas otras, comensal y puede ser aislada tanto de piel como de mucosas en perros y humanos. En el 2016 se aisló y secuenció el genoma completo de la cepa SW120 de S. cohnii subsp. urealyticus aislada a partir de un hisopado de oído de un perro (Bean et al., 2017). Al igual que una de nuestras cepas de S. conhii aisladas, esta cepa SW120 presenta el fenotipo resistente a meticilina.
Trabajos realizados en los Países Bajos en el año 2004 indicaron que un 36% de las cepas de S. haemolyticum aisladas de perros y gatos fueron resistentes a meticilina (van Duijkeren et al., 2004). Por otro lado, en Lituania en un trabajo realizado por Ruzauskas y colaboradores en el año 2014, se obtuvieron un 1,6% de resistencia en aislamientos de S. haemolyticus obtenidos de perros (Ruzauskas et al., 2014). Posteriormente, en otro trabajo realizado McManus y colaboradores obtuvieron un total de 20 cepas de S. haelolyticum resistentes en un total de 21 cepas de esta bacteria aislada de perros, estos valores representan una frecuencia de resistencia de 95,2% (McManus et al., 2015). Nuestro muestreo permitió el aislamiento de dos cepas de S. haemolyticus resistentes a meticilina de un total de siete cepas identificadas de esta especie, representando un porcentaje menor a lo reportado previamente (28% aproximadamente). Podemos decir que en los linajes circulantes de S. haemolyticum en Montevideo existe el potencial para adquirir genes de resistencia a meticilina, de hecho, varias de las cepas cercanamente relacionadas han sido descritas como resistentes. Por otro lado, el menor porcentaje obtenido en este estudio podría sugerir también que los genes de resistencia están, se han adquirido recientemente en estas cepas o que estamos frente a una estabilidad en cuanto a la resistencia, que sería disparada con el potencial uso excesivo de los β-lactámicos. Otra posibilidad es que el bajo valor de resistencia encontrado sea una subestimación, por lo que sería necesario un muestreo más grande para lograr observar cómo se comporta la resistencia en las cepas de esta especie.
S. arlettae fue aislado por primera vez en el año 1984 por investigadores alemanes. Estos aislamientos provenían de muestras de piel y faringe de aves de corral y cabras (Schleifer et al., 1984). Posteriormente se determinó que esta especie tenía el potencial de degradar colorantes utilizados en la industria textil y del cuero (colorante azóicos), así como también algunas cepas tenían la capacidad de promover el crecimiento vegetal. Solamente uno de nuestros aislamientos fue una cepa de S. arlettae, éste provenía de hisopado de faringe. El aislamiento reportado en este trabajo representa el primero de la especie en perros. No es claro, por lo tanto, qué potencial patógeno podrían tener en perros u otros animales domésticos.
Resistencia a antimicrobianos en Staphylococcus
La resistencia a penicilina encontrada en el total de aislamientos fue de un 75% lo que indica que, en aislamientos de animales, a pesar de tener una amplia presión de selección por parte de este antibiótico, todavía circulan cepas sensibles.
En el año 2015 en Lituania un grupo de investigadores realizó un relevamiento de los patrones de resistencia en Staphylococcus aislados a partir de mascotas. En el trabajo los autores reportan que la resistencia a meticilina estaba mayormente concentrada en cepas de la especie de S. pseudintermedius al igual que ocurrió con nuestros aislamientos (de las siete resistentes tres eran S. pseudintermedius). Cabe pensar que al ser la especie mayoritaria en los perros dentro de los Staphylococcus tiende a acumular mayor cantidad de genes de resistencia en su genoma, por lo que la frecuencia de resistencia se vincularía mayormente a su mayor presencia y a su asociación histórica con el hospedero (Ruzauskas et al., 2015).
La resistencia a vancomicina, por otro lado, fue nula en las cepas aisladas de perros, lo que coincide con lo encontrado en trabajos de otros países como Lituania (Ruzauskas et al., 2015) y Turquía (Aslantas et al., 2013). La técnica de antibiograma aquí empleada detecta un tipo de resistencia a vancomicina que es la resistencia absoluta. Las cepas de Staphylococcus con este tipo de resistencia son muy poco frecuentes lo que se ve reflejado en nuestros resultados donde no se obtuvo ninguna. El problema fundamental en la clínica con respecto al uso de este antibiótico es la aparición de cepas con resistencia intermedia a vancomicina, las denominadas cepas VISA (vancomycin-intermediate S. aureus) que no pueden ser detectadas por antibiograma.
La resistencia a eritromicina encontrada fue de un 24%, un valor pequeño si lo comparamos con los reportados en el trabajo de Ruzaskas en el 2015 que obtuvo un 87,5% de resistencia a este antibiótico.
En Brasil el porcentaje de resistencia a eritomicina detectada fue de un 57% (de Godoy et al., 2016) y en Corea fue de un 64% (Jang et al., 2014). El bajo porcentaje obtenido a partir de nuestros aislamientos podría indicar que la presión selectiva frente a este antibiótico es mucho mayor en los países nombrados que en el nuestro, ya que el uso de eritromicina está menos extendido y no es de uso habitual en la clínica veterinaria.
La resistencia a kanamicina detectada también fue más baja en comparación con trabajos previamente reportados. En Portugal en el año 2016 se observó que en cepas aisladas de perros la resistencia a kanamicina era de un 19% (Couto et al., 2016). Solamente un 10% de nuestras cepas aisladas resultaron resistentes a este antibiótico.
Con respecto a la resistencia a enrofloxacina, el porcentaje de cepas obtenido fue de un 18%, también menor a lo reportado en Corea (42,3%) y similar a lo reportado en Portugal (Couto et al., 2016; Jang et al., 2014).
Es muy importante destacar que en nuestro país el uso de la enrofloxacina en medicina veterinaria está muy extendido, y va en aumento, para tratar cualquier tipo de lesión de piel, otitis, etc. No existen reportes publicados anteriormente sobre la prevalencia de la resistencia a este antibiótico en nuestro país en pequeños animales. Por conocimientos previos del laboratorio del área en cuanto al procesamiento de muestras clínicas, se ha visto de un tiempo a esta parte que las cepas resistentes a enrofloxacina han ido en aumento. Por otro lado, es importante notar que este antibiótico es utilizado exclusivamente a nivel veterinario por lo que podemos deducir que la resistencia al mismo ha aparecido y se ha expandido en cepas exclusivamente de animales domésticos.
Con respecto a la resistencia frente a un antibiótico combinado con un inhibidor de ß-lactamasa, como por ejemplo amoxi/clavulánico, se obtuvo un 10% de resistencia al igual que la meticilina. Las cepas que fueron resistentes a meticilina y presentaban el gen mecA eran resistentes también al resto de los β-lactámicos, incluyendo la amoxicilina clavulánico como indica la CLSI. Este resultado es similar con el reportado en Turquía, Corea y Portugal (Aslantas et al., 2013; Couto et al., 2016; Jang et al., 2014).
Conclusiones
A partir de las muestras tomadas de diferentes perros que ingresaron al Centro hospitalario de la Facultad de Veterinaria en el período de mayo 2016 a mayo de 2017 se lograron obtener 67 aislamientos. Se identificaron en su totalidad por análisis fenotípico y por secuenciación del ARNr 16S, determinando que las especies que se encuentran circulando en los perros son: S. epidermidis, S. arlettae, S. pseudintermedius, S. xylosus, S. nepalensis, S. argenteus, S. cohnii, S. haemolyticus y S. schleiferi.
Siete de estos 67 aislamientos resultaron ser resistentes a meticilina y contenían el gen mecA y 10 resultaron ser cepas MDR (resistentes a multiple drogas).
Finalmente podemos concluir que las condiciones para la transferencia de genes de resistencia a meticilina en caninos están dadas ya sea por la existencia de dichos genes y los elementos genéticos móviles en los que ellos son transportados, así como también por la presencia de cepas bacterianas capaces de incorporarlos.