Introducción
Los recientes avances en genética molecular han hecho posible la identificación en forma eficiente y rápida de animales heterocigotos mediante análisis genómico. Conocer la base molecular de un defecto posibilita la detección directa de los portadores a nivel genético (Agerholm, 2007). En la actualidad se conocen alrededor de 246 enfermedades genéticas relacionadas con todas las especialidades veterinarias en el ganado bovino, la mayoría de ellas son del tipo monogénico (OMIA, 2018). BLAD (deficiencia en la adhesión leucocitaria bovina, 000595-9913), DUMPs (deficiencia en la enzima uridina monofosfato sintasa, 000262-9913) y Citrulinemia (deficiencia en la enzima arginosuccinato sintetasa, 000194-9913) son enfermedades de herencia autosómica recesiva y han sido descritas en el catálogo OMIA, en el cual está recopilada toda la información sobre estas tres enfermedades con las mutaciones reportadas por sus autores (Shuster y col., 1992; Schwenger y col., 1993; Dennis y col., 1989; respectivamente).
Se han utilizado tests genéticos que permitieron identificar las mutaciones de estas tres enfermedades en poblaciones de animales Holando en Uruguay tales como: PCR-RFLP (del inglés Polymerase Chain Reaction - Restriction Fragment Length Polymorphism) (Llambí, 2002; Llambí y col., 2003; Kelly y col., 2010; Branda Sica y col., 2016), el análisis del electroferograma de la secuencia del amplicón (Branda Sica y col., 2016) y PCR-HRM (con la aplicación High Resolution Melting) (Branda Sica y col., 2015; Federici y col., 2018). A partir del año 2007 se desarrollaron tecnologías de genotipado en paneles que permiten evaluar de forma simultánea una gran cantidad de SNPs (siglas en inglés, Single Nucleotide Polymorphism) distribuidos de manera uniforme en todo el genoma bovino. Existen paneles de diferentes densidades, el de baja densidad que permite evaluar 6.909 SNPs de forma simultánea (IlluminaSNP7K), el de alta densidad, el “BovineHD” que cuenta con sondas específicas para evaluar 777.962 SNPs diferentes y el de media densidad ó 50K (BovineSNP50) que incluye sondas para 54.600 SNPs altamente informativos distribuidos de manera uniforme en todo el genoma bovino. Dado que el costo de genotipar un animal con un panel de más de 50.000 SNPs era demasiado alto para su uso masivo en la población se desarrollaron los paneles de baja densidad para estudios específicos. El primer panel de baja densidad, el Bovine3K con 3.072 SNPs fue desarrollado y comercializado por la empresa Illumina y USDA-ARS en 2010. Con el desarrollo de métodos de imputación de genotipos, el genotipado con paneles de baja densidad (BovineLD y GGP) se ha incrementado, pues en comparación con paneles de media densidad presentan una muy pequeña reducción en la exactitud, pero aproximadamente a la mitad del costo, siendo una opción eficiente para el genotipado de poblaciones grandes (Wiggans y col., 2013). Los paneles de baja densidad utilizados para ganado de leche son: GeneSeekDairy-ULTRA-LDv2 (7.049 SNPs) y los GGP-BovineLDv1, v2, v3 y v4 con 8.610, 19.721, 26.151 y 30.125 SNPs, respectivamente, de GeneSeek (Neogen Corporation), y los BovineLDv1 (6.912 SNPs) y BovineHD777K de Illumina. En este estudio se utilizó el panel GGP-BovineLDv3 que incluye la detección de los SNPs asociados a las enfermedades monogénicas BLAD, Citrulinemia y DUMPs que antes venían como tests individuales.
Un tema no menor es la consideración de los costos de las técnicas moleculares tradicionales de PCR y sus variantes frente al uso de un panel de baja densidad como el utilizado en el presente trabajo (GGP-BovineLDv3).
El objetivo de este trabajo consistió en realizar un screening de las enfermedades hereditarias BLAD, DUMPs y Citrulinemia, en una población representativa de terneras Holando de la región este utilizando el panel GGP-BovineLDv3 de 26K y comparar la evolución temporal de estas enfermedades en el país (2003 al 2018). Se validaron las técnicas previamente utilizadas en la muestra poblacional de terneras Holando de recría reportados por Branda Sica y col. (2016 y 2015).
Materiales y métodos
Muestras y material de referencia
Se colectó sangre de 190 terneras de recría (cohortes representativas de 24 predios de la cuenca lechera de Cerro Largo, Uruguay). Estas terneras de recría fueron previamente analizadas para la detección de alelos mutantes de BLAD y Citrulinemia mediante PCR-RFLP, y PCR- secuenciación (Branda Sica y col., 2016), y PCR en tiempo real con la aplicación HRM para BLAD (Branda Sica y col., 2015).
Extracción de ADN, concentración y calidad de ADN
El ADN fue extraído a partir de glóbulos blancos siguiendo las normas y los protocolos del Banco de ADN Genómico Animal de la Unidad de Biotecnología, INIA Las Brujas, versión modificada del protocolo de Green y Sambrock (2012). Se determinó la concentración del ADN por nanodrop a 260 nm (NanoDrop 8000 spectrophotometer, Thermo Scientific) y su calidad por la relación OD260/OD280.
Análisis genético de los datos
Estas muestras de ADN fueron enviadas a genotipar a la empresa Neogen Corporation de GeneSeek, donde se realizó el genotipado con el panel GGP-BovineLDv3 de 26K, de análisis químico Infinium de plataforma Illumina. Para identificar si las terneras Holando son portadoras de las enfermedades hereditarias: BLAD, Citrulinemia y DUMPs, se consideraron los genotipos con call rate superior a 90 % asegurando la alta calidad de los datos genómicos en esta población analizada. Las frecuencias alélicas y genotípicas fueron calculadas por conteo directo según el método de Nei (1989).
Resultados
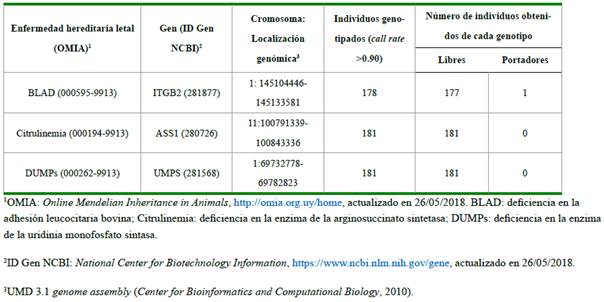
Mediante el genotipado con el panel GGP-BovineLDv3 de 26.151 SNPs se detectó la presencia del alelo mutante para BLAD en una sola ternera de recría en un total de 178 terneras genotipadas (call rate >0.90). La frecuencia del alelo mutante para BLAD en esta muestra poblacional estudiada fue baja, de q=0.0028, y la frecuencia genotípica de terneras portadoras de BLAD, 0.56 %. No se encontraron alelos mutantes para DUMPs y Citrulinemia en un total de 181 terneras genotipadas, respectivamente (call rate >0.90). En el Cuadro 1 se muestra el número total de animales genotipados utilizando dicho panel, así como de genotipos normales y portadores de BLAD, Citrulinemia y DUMPs, con sus identificaciones respectivas en el genoma bovino. En la Figura 1 se muestra un gráfico de comparación de la evolución a través del tiempo de la frecuencia del alelo mutante de la enfermedad BLAD en los distintos relevamientos realizados en Uruguay.
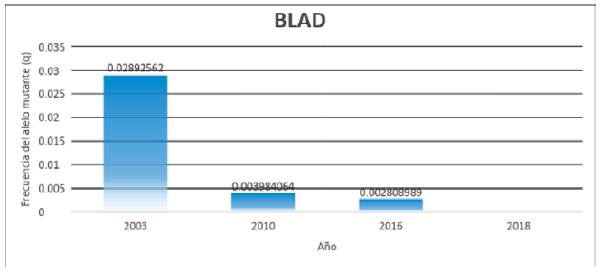
Figura 1: Evolución de las frecuencias del alelo mutante (q) de la enfermedad BLAD en Uruguay analizadas según los estudios de Llambí y col. (2003); Kelly y col. (2010); Branda Sica y col. (2016); y Federici y col. (2018).
Cuadro 1: Número de individuos genotipados libres y portadores de enfermedades hereditarias letales utilizando el panel GGP-BovineLDv3, 26 K (GeneSeek Genomic Prolifer de baja densidad para bovino, versión 3, con 26.151 SNPs), los genes asociados con su identificación en NCBI y su localización en el genoma
Discusión
Se compararon los resultados del genotipado utilizando el panel GGP-BovineLDv3 de 26.151 SNPs con los 2 protocolos de PCR-RFLP (para BLAD y Citrulinemia), y 2 de confirmación por PCR-secuenciación (BLAD y Citrulinemia) y 1 de PCR-HRM (BLAD), respectivamente estudiados en los trabajos de Branda Sica y col., 2016 y 2015, en la muestra poblacional de terneras de recría. El 100 % de concordancia entre los resultados de los cinco métodos indican tanto la alta sensibilidad y especificidad del panel GGP-BovineLDv3. La frecuencia de terneras portadoras de BLAD (0.56 %) fue menor a las reportadas previamente por Llambí y col. (2003); y Kelly y col. (2010) (6.6 % y 0.79 %, respectivamente) Con respecto a la evolución de la frecuencia del alelo mutante de BLAD desde su primer reporte en 2003 al presente en Uruguay, se observó una importante disminución (Llambí y col., 2003; Kelly y col., 2010; Branda Sica y col., 2016; Federici y col., 2018) (Figura 1). Esta disminución podría deberse al uso de semen genotipado libre de estas enfermedades en los últimos años. La alta frecuencia en el muestreo reportado por Llambí y col. (2003) refleja el hecho de que fueron seleccionadas muestras de tamaño pequeño en el que se encontró que fue utilizado como reproductor un toro portador de BLAD. Estos cuatro trabajos representan las únicas fuentes de información sobre animales portadores de BLAD mediante técnicas de diagnóstico molecular en Uruguay y éste sería el primer estudio comparativo de la evolución de este alelo mutante en el tiempo desde que se detectó esta enfermedad por primera vez en 2003. En Alemania, Schütz y col. (2008) encontraron que la frecuencia del alelo mutante para BLAD disminuyó aproximadamente a 1,6 % en 2007 en una población de vacas Holando que fueron genotipadas utilizando dos tests genéticos como PCR-RFLP y PCR-FRET, y se demostró que mediante la selección asistida por marcadores se podía reducir considerablemente la frecuencia de una mutación dentro de un período no más de 5 años. La incidencia de BLAD fue muy elevada (23 %) en estudios anteriores a partir del primer reporte de Shuster y col. (1992). Debido a los análisis de screening regulares, las incidencias se están reduciendo en todo el mundo (Meydan y col., 2010).
Todos los animales validados en este estudio presentaron un genotipo homocigota normal para la enfermedad DUMPs, no identificándose alelos mutantes en la población analizada. Esta enfermedad había sido detectada en EE.UU y Europa (Robinson y col., 1993) por la importación de semen de “Happy Herd Beautician”, uno de los mejores toros Holando de EE.UU en 1987. En Uruguay no hay reportes de presencia de portadores de la enfermedad DUMPs desde 2002 (Llambí, 2002; Kelly y col., 2010). Tampoco se encontraron animales portadores de Citrulinemia en los reportes de Llambí (2002); Kelly y col. (2010); Meydan y col. (2010); y Branda Sica y col. (2016). Esta enfermedad había sido detectada en Australia, EE.UU y Europa por la importación de semen de un toro Holando de pedigrí conocido mundialmente como “Linmarck Kriss King” de EE.UU (Healy y col., 1991).
Se recomienda el screening utilizando paneles de baja densidad, como el GGP-BovineLDv3, en toros de cría como control para prevención y monitoreo de los alelos mutantes en relación con las técnicas moleculares previamente citadas dada la gran cantidad de información aportada por este panel, la alta precisión en el genotipado de SNPs vinculados a enfermedades hereditarias y actual tendencia a la disminución de los costos por muestra/animal. Debemos tener en cuenta que la utilización de técnicas tradicionales de PCR y sus variantes (PCR- RFLP, PCR- secuenciación y PCR-HRM) tienen un costo estimado/muestra de U$s 39, 40 y 15, respectivamente, frente al panel de baja densidad, U$s 40/muestra.
Considerando la calidad de información genómica y cantidad de SNPs vinculados a enfermedades que se están incorporando junto a los caracteres productivos genotipados en un solo test de ADN, este insumo puede ser de gran utilidad al momento de tomar decisiones y establecer estrategias de cría, apoyando el mantenimiento de la salud de los terneros y la sostenibilidad de la raza Holando.











 Curriculum ScienTI
Curriculum ScienTI