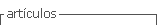Introducción
Testeo, testeo, testeo, ha sido la recomendación de la Organización Mundial de la Salud (OMS) frente a la pandemia de COVID-19. Más de 500 test o pruebas diagnósticas se están comercializando en el mercado mundial o se encuentran en avanzada fase de desarrollo1. La falta de un estándar de oro, el uso de diferentes métodos de recolección y preparación de muestras, y una comprensión, aún incompleta, de la dinámica viral en el curso de la infección, dificultan la evaluación rigurosa de la precisión diagnóstica de los innumerables ensayos de SARS-CoV-2 recientemente introducidos2.
En esta situación, sin precedentes, es importante comprender el tipo de ensayos disponibles así como su rol en el proceso diagnóstico. Y lo más crítico, en términos de la seguridad del paciente, es no asumir que todos los ensayos son siempre precisos en este escenario de una infección emergente3.
Cuando hablamos de COVID-19 debemos manejar tres premisas:
1. estamos frente a un virus de muy reciente descripción,
2. se trata de una pandemia sin precedentes en los tiempos modernos y
3. estamos trabajando en territorio desconocido y con bibliografía que, muchas veces y hasta este momento, se publica sin revisión de pares3.
Metodología
Para la elaboración del presente trabajo revisamos la bibliografía publicada en formato web y disponible en PubMed, o en los portales de cada publicación. También tomamos en cuenta las recomendaciones realizadas por los siguientes organismos: Organización Mundial de la Salud (OMS), Organización Panamericana de la Salud (OPS), Centers for Disease Control and Prevention (CDC), European Centre for Disease Prevention and Control (ECDC), Foundation for Innovative New Diagnostics (FIND), Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) y Ministerio de Salud Pública del Uruguay (MSP). Se consideraron las publicaciones realizadas hasta el 30 de junio de 2020.
Desarrollo y discusión
Concepto de sensibilidad y especificidad analítica y clínica
Vamos a abordar los conceptos de sensibilidad y especificidad analíticas así como los de sensibilidad y especificidad clínicas. La sensibilidad analítica es la cantidad más pequeña de sustancia que se puede detectar en una muestra, de manera confiable, mediante un ensayo (límite de detección). En términos prácticos, para medir la sensibilidad analítica de la prueba molecular para SARS CoV-2 se determina la cantidad mínima de ácido ribonucleico (ARN) que la prueba detectará 95 de cada 100 veces. La especificidad analítica es la capacidad del ensayo para detectar sólo la sustancia deseada en una muestra sin reacción cruzada con otras sustancias. Sin embargo, estas definiciones difieren con los conceptos de sensibilidad y especificidad clínica (el porcentaje de pacientes enfermos que dan positivo y no enfermos que tienen resultados negativos, respectivamente). Una prueba con buena sensibilidad y especificidad analítica no necesariamente tiene buena sensibilidad y especificidad clínica4. Lamentablemente, el rendimiento clínico de las pruebas moleculares para detectar SARS-CoV-2 no puede conocerse hasta que comprendamos quién está realmente infectado y quién no.
Hasta el momento el diagnóstico de laboratorio de COVID-19 se puede realizar mediante tres métodos. El recomendado por todas las agencias internacionales, e incuestionable, es la detección molecular de ARN viral en muestras respiratorias5,6. Luego encontramos los métodos rápidos de detección de antígeno del virus en muestras respiratorias y, por último, los métodos serológicos que buscan la respuesta de anticuerpos de tipo IgM, IgA, IgG o totales. Los diversos métodos presentan diferente sensibilidad y especificidad clínica. A su vez, en el proceso diagnóstico, esta performance depende de la calidad de la muestra y del día en que la misma se colecta, en relación al día de inicio de los síntomas.
Detección molecular de SARS-CoV-2
Como se citó anteriormente, la utilización de la detección molecular del ARN viral como prueba diagnóstica es incuestionable, dado su excelente desempeño analítico. Cabe destacar que la sensibilidad y especificidad analíticas de la mayoría de las técnicas de retrotranscripción seguida de reacción en cadena de la polimerasa (RT-PCR) son excelentes7.
Sin embargo, cuando se considera el proceso diagnóstico completo partiendo desde la recolección de la muestra, la sensibilidad clínica desciende en función del tipo y la calidad de muestra, el transporte y el almacenamiento de ésta, así como la etapa evolutiva de la enfermedad8,9.
Para las pruebas de diagnóstico molecular inicial de SARS-CoV-2, el CDC de Estados Unidos recomienda colectar y analizar muestras del tracto respiratorio superior. Estas muestras comprenden el hisopado nasofaríngeo y/o orofaríngeo y/o nasal, presentando el primero mejor sensibilidad y siendo el de preferencia5. Se pueden utilizar otras muestras como el aspirado nasofaríngeo, expectoración, aspirado traqueal y lavado bronquioloalveolar5,6.
Preanalítica
El proceso diagnóstico comienza con la indicación del estudio. Sin entrar en los distintos enfoques para indicar los estudios, entendemos que la indicación debe ser pautada por un consenso entre médicos prescriptores y profesionales del laboratorio clínico. Particularmente, en esta pandemia, han realizado un importante aporte los grupos de expertos Intercátedra de la Universidad de la República10, así como el MSP cumpliendo este último un rol importante en la articulación, coordinación y definición de pautas.
Recolección de muestra
Este procedimiento, como fue citado, tiene un rol preponderante en el éxito de la prueba diagnóstica, por lo tanto su realización deberá cumplir con los mejores estándares de calidad. Para la recolección adecuada de la muestra debemos plantearnos tres elementos importantes.
1. Oportunidad. El virus del SARS-CoV-2 puede detectarse inicialmente en muestras de HNF uno a dos días antes del inicio de los síntomas11,12 y persiste durante siete a doce días en casos moderados13. Sin embargo, esto depende del curso clínico de la enfermedad, habiéndose reportado la persistencia de prueba positiva durante más de veinte días13. En pacientes graves se recomienda realizar toma de muestra de tracto respiratorio profundo si el primer estudio de HNF fue negativo14.
2. Calidad de la muestra. El HNF tiene un protocolo de recolección bien establecido y es un procedimiento molesto para el paciente, incluso puede provocar lagrimeo15. No se puede usar cualquier hisopo. Debe ser flexible y su punta debe ser de poliéster con eje de material plástico. No está recomendado el uso hisopos de alginato de calcio, algodón o hisopos con ejes de madera, ya que pueden contener sustancias que inactivan virus o inhiben las pruebas moleculares16.
3. Transporte y almacenamiento. El hisopo obtenido se debe colocar en un tubo, de preferencia, con medio de transporte viral (MTV). Las alternativas para el transporte serían: soluciones tamponadas (PBS) o suero fisiológico (SF)17. Si se colecta muestra orofaríngea (OF), ambos hisopos (HNF y OF) deben combinarse en un solo tubo con líquido de transporte. Al colocar los hisopos dentro de los tubos, se debe cortar el eje del hisopo, utilizando hisopos troquelados, o a través del uso de material cortante estéril o adecuadamente desinfectado entre recolecciones.
El aspirado nasofaríngeo (ANF) está especialmente indicado en niños menores de 2 años. Este se realiza con una sonda asociada a un frasco o tubo trampa. En este caso, se puede enviar el aspirado si se prevé un procesamiento antes de las 24 horas de recolectada la muestra, pero es recomendable luego de realizar el ANF propiamente dicho, aspirar medio de transporte viral al tubo trampa. De una manera similar se haría con otras muestras, como el aspirado traqueal o lavado bronquioloalveolar, indicados en pacientes sometidos a ventilación mecánica invasiva4. Las muestras colectadas de esta manera no pueden ser compartidas para uso de detección de otros virus respiratorios, salvo se efectúe en el propio laboratorio donde se realice las búsqueda de SARS-CoV-2 y se realicen alícuotas en forma segura.
Una vez que tenemos la muestra en el tubo, debemos asegurarnos que esté rotulado con los datos filiatorios del paciente. Muchos laboratorios utilizan etiquetas con código de barras. Para el transporte interno, el tubo se debe colocar en doble embolsado, mediando antisepsia de manos entre embolsado primario y secundario. Este transporte debería realizarse en recipiente rígido, cerrado con tapa, manteniendo el tubo en posición vertical. En el caso de muestras que deben salir fuera de la institución se recomienda el triple envase, reglamentado en el anexo 4 del decreto 129/00518. Las muestras deben transportarse al laboratorio a una temperatura entre 2 ºC y 8 °C, destacando que la la muestra debería procesarse antes de las 72 horas desde recolección, si se utiliza medio de transporte viral y dentro de las 24 horas de colectada si se utiliza SF o PBS5. Sin el medio de transporte o almacenamiento adecuado, las muestras se degradan. Esto es especialmente cierto para el ARN que se detecta mediante una prueba de RT-PCR. El ARN es menos estable que el ácido desoxirribonucleico (ADN), por lo que si una muestra no se transporta o almacena adecuadamente, aumenta el riesgo de un resultado RT-PCR falso negativo.
Una vez que la muestra llega al laboratorio, se realizan los controles habituales preanalíticos de la muestra. La conservación dentro del laboratorio también debe realizarse a temperatura entre 2 ºC y 8 °C hasta poder iniciar el procesamiento. El procesamiento inicial (antes de la inactivación) de todas las muestras debe realizarse en cabina de bioseguridad, debidamente mantenida y validada19.
Analítica
El procedimiento analítico dependerá de las características del laboratorio, de la capacitación del personal así como del equipamiento existente. La detección molecular comprende dos procedimientos: la extracción del ARN y posteriormente RT-PCR.
Extracción de ARN
Esta parte del proceso analítico define, en muchas ocasiones, las características del flujo de trabajo del procedimiento completo. En base al equipamiento de extracción de ARN, podemos clasificar a los equipos automatizados en equipos de baja, mediana y alta capacidad de procesamiento en función de la cantidad simultánea de muestras capaz de procesar. Los equipos de baja capacidad presentan en general una respuesta rápida (aproximadamente tres horas) para un bajo número de muestras (una a cuatro muestras); sin embargo, los equipamientos de alta capacidad presentan un tiempo de procesamiento más prolongado (aproximadamente ocho horas) pero con número de muestras procesadas significativamente mayor (96 muestras). En general, en este último grupo el procesamiento se realiza en tandas, lo que implica juntar una determinada cantidad de muestras para luego iniciar el proceso analítico. Ante la carencia de insumos para algunos equipos de extracción de ARN automatizados, cabe señalar como una alternativa válida la utilización de procedimientos de extracción manual, aunque este tipo de metodología debe enmarcarse en un riguroso sistema de trazabilidad, capacitación del personal técnico así como la utilización de kits comerciales validados para este fin.
RT-PCR
Este proceso comprende la etapa de retrotranscripción (RT) y la etapa de amplificación. La RT puede realizarse de forma separada o unida en un mismo paso a la segunda, según el protocolo o kit utilizado. Para comprender los diferentes protocolos utilizados en la etapa de la amplificación de SARS-CoV-2 debemos comprender el contenido genético del virus, el cual se observa en la (figura 1). Desde el inicio de la circulación e identificación del SARS-CoV-2 se comenzaron los trabajos de puesta a punto de protocolos para la detección del virus en diferentes centros académicos y científicos del mundo. La OMS realizó una evaluación y recomienda la utilización de los protocolos que se observan en la (tabla 1).
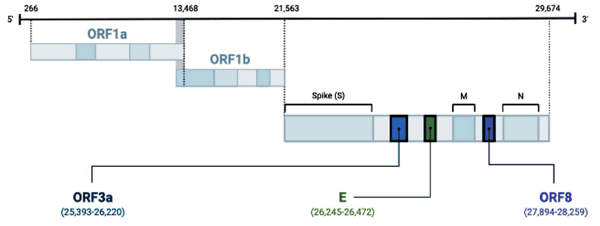
Figura 1: Genes de SARS-CoV-2 y dianas moleculares utilizadas en los protocolos moleculares. El genoma codifica dos grandes genes ORF1a y ORF1b que codifican 16 proteínas no estructurales, dentro de las cuales se encuentra la RNA polimerasa-RNA dependiente (RdRp). Los genes estructurales codifican la proteína Spike o espícula (S), la de envoltura (E), la de membrana/matriz (M) y la de nucleocápside (N).
Los protocolos citados presentan diferentes objetivos moleculares, y a su vez los mismos tienen pequeñas diferencias en su sensibilidad analítica20. Sin embargo, todos muestran un muy buen desempeño para el diagnóstico de este virus. Si el límite de detección (LoD) para una prueba es demasiado alto, los pacientes infectados con SARS-CoV-2 pueden no dar positivo, lo que lleva a una alta tasa de resultados falsos negativos. Si el LoD para una prueba es demasiado bajo, entonces la contaminación puede convertirse en un problema importante, ya que la prueba detectará las cantidades más pequeñas de ARN viral, lo que conducirá a resultados positivos falsos (es decir, personas que no están infectadas con SARS-CoV- 2 darán positivo en lugar de negativo). El LoD está disponible en las instrucciones de uso de las pruebas de SARS-CoV-2 comerciales.
Aunque la recomendación para la confirmación de caso establece la detección de dos objetivos moleculares positivos (por ejemplo, N y RdRp o E y RdRp), la OPS ha recomendado que, una vez establecido el virus en la región o país, es suficiente la detección de un solo objetivo molecular para confirmar el diagnóstico9. Cabe advertir que si bien la mayoría de los objetivos moleculares presentan alta sensibilidad analítica, RdRp tiene una sensibilidad algo menor al resto, por lo que su utilización como único objetivo molecular debe desaconsejarse20). En Uruguay, investigadores de la Universidad de la República y del Instituto Pasteur desarrollaron una prueba de RT-PCR21. Esta prueba es comercializada por la empresa Atgen y detecta secuencias específicas del gen que codifica para la proteína N de la nucleocápside viral22.
Posanalítica
La interpretación de las pruebas moleculares se debe hacer con prudencia. A pesar de que se han revelado algunas correlaciones, las cargas virales determinadas por RT-PCR no deben utilizarse para indicar la gravedad de la infección o para controlar la respuesta terapéutica2,11,23. Sin embargo, valores bajos de Ct (umbral de ciclo) indican altas cargas virales y pueden usarse como una indicación de mayor transmisibilidad24. Ct es la intersección entre una curva de amplificación y una línea de umbral. Es una medida relativa de la concentración del objetivo en la reacción de PCR en tiempo real.
La interpretación de los resultados se debería realizar valorando el contexto clínico-epidemiológico que presenta el paciente, especialmente en aquellos que requieran internación. Los resultados negativos en un escenario de alta sospecha deben obligarnos a valorar las posibles causas de falsos negativos. Pueden aparecer falsos negativos en las siguientes circunstancias8,9.
- Recolección inadecuada del HNF.
- Retraso o pérdida de cadena de frío durante el transporte.
- Error preanalítico en el etiquetado de la muestra a lo largo del proceso.
- Poca eliminación de virus por el paciente dado el estadio de la infección o por la gravedad de ésta.
- Presencia de mutaciones del virus en las regiones a las que se dirigen los ensayos.
Además de estos aspectos preanalíticos también puede haber factores analíticos relacionados con la aparición de falsos negativos. En ese sentido, la sensibilidad podría verse afectada por mutaciones virales en los sitios objetivo, así como por la presencia de inhibidores presentes en la muestra. Ambas situaciones, hasta el momento, fueron muy poco frecuentes. Sin embargo, en un estudio que analizó 373 secuencias genéticas de virus SARS-CoV-2 de Latinoamérica, incluyendo 11 de Uruguay, se demostró la presencia de cambios en las regiones del gen N que podrían impedir un diagnóstico molecular preciso25. En cambio, la región del gen E parece mantenerse conservada, por lo que los autores la recomiendan como candidata ideal para la detección de primera línea en Latinoamérica25. En Europa, por el contrario, ya se ha detectado falla en la detección del gen E debido a una mutación26. Por tanto, a pesar de las recomendaciones de la OPS de investigar un objetivo único, a la luz de estos trabajos resulta riesgoso confiar en un ensayo dirigido a una única secuencia del genoma viral. También se pone de manifiesto la necesidad de mantener la vigilancia, a través de secuenciación completa, de SARS-CoV-2 con el objetivo de buscar variantes que interfieran con la capacidad de detección de las pruebas de RT-PCR. Finalmente, sería recomendable que los fabricantes fueran transparentes sobre los cebadores y las sondas utilizadas, ya que esto permitiría que las variantes problemáticas se identificaran más fácilmente a partir de las secuencias virales disponibles.
En los ensayos moleculares el hallazgo de falsos positivos es muy poco frecuente. Estos podrían deberse a error preanalítico o contaminación cruzada entre muestras durante el procesamiento analítico8.
Luego de la interpretación de los resultados, se realiza el informe escrito con los resultados obtenidos y la firma del profesional que se responsabiliza por los resultados27.
Detección de antígenos virales
Al día de estar realizando este manuscrito, estas pruebas no están disponibles en nuestro país. Sin embargo, el desarrollo de anticuerpos monoclonales contra la proteína de nucleocápside es la base de los métodos rápidos de detección de antígenos de SARS CoV-2 en muestras respiratorias28.
En un estudio, aún sin revisión de pares, este tipo de ensayo mostró una sensibilidad de 50% cuando la RT-PCR fue positiva y de 82,2% frente a un Ct < 25. La especificidad reportada fue de 100%29. La SEIMC emitió recomendaciones señalando que los ensayos preliminares realizados con este tipo de pruebas, disponibles en España, mostraron una sensibilidad inferior a 30% y una especificidad de 100%. La Sociedad afirmó que estos resultados impedirían su introducción en la rutina8. Por otra parte, desconocemos la cinética de detección de antígenos, presuponemos que sería similar a la del ARN viral9. Por tanto, a pesar de los prometedores resultados de algunos trabajos, cualquier tipo de prueba de este tipo que se fuera a comercializar debería pasar por un proceso de verificación con muestras respiratorias. Si se verifica su alta especificidad podría utilizarse en aquellos pacientes sintomáticos que requieran internación. Por otro lado, dada la sensibilidad reportada, solo sería de utilidad en aquellos casos que diera positiva. Es claro que si esta prueba da negativa, se debe realizar el ensayo molecular.
Las ventajas de este tipo de ensayo es que se trataría de pruebas rápidas que se podrían realizar en cualquier laboratorio en condiciones de bioseguridad adecuadas8.
Detección de respuesta inmune
Introducción
Generalmente en las infecciones virales, alrededor del inicio de los síntomas, los leucocitos inician el proceso de fabricar anticuerpos contra diferentes estructuras virales. Al principio estos anticuerpos, de tipo IgM, son bastante inespecíficos, lo cual podría dar lugar a reacciones cruzadas. Estos anticuerpos son transitorios y se tornan indetectables luego de algunas semanas. Los anticuerpos de tipo IgA, característicos de las secreciones mucosas, tienen una cinética de aparición similar a los IgM30. A lo largo del tiempo la respuesta inmune madura, se hace más específica contra antígenos propios de SARS CoV-2. Igual hay posibilidad que estos anticuerpos de tipo IgG, aún, reaccionen contra antígenos presentes en la misma familia de virus. Sin embargo, la aparición de IgG indica que el paciente se ha infectado con el virus y ha montado una respuesta inmune contra éste. Solo una publicación realizada en modelo animal y aún sin revisión de pares, establece que la respuesta inmune protege contra la reinfección31. Por tanto, aún falta evidencia para afirmar que la respuesta inmune es protectora.
Pruebas serológicas
Las pruebas serológicas que identifican IgA, IgM, IgG o incluso anticuerpos totales contra SARS-CoV-2 se realizan en muestras de sangre, suero o plasma. Estas pruebas son menos complejas de realizar que las pruebas moleculares. Sin embargo, no son útiles para diagnosticar infecciones en etapa aguda32. La respuesta de anticuerpos a la infección tarda un par de semanas en ser detectables de manera confiable30. Los resultados negativos no excluyen la infección por SARS-CoV-2, particularmente en pacientes con exposición reciente al virus. Existen estudios publicados que utilizaron sustratos antigénicos variados y elaborados en cada laboratorio. Estos incluyeron: proteína de nucleocápside (N)29, combinación de proteína N más proteínas de espícula23 y otros utilizaron solo proteínas de espícula32.
Para valorar la especificidad de los ensayos serológicos, el estudio de Guo y colaboradores30 utilizó plasmas con anticuerpos contra los coronavirus humanos -229E, -NL63, -OC43, -HKU1, y SARS-CoV. La prueba serológica para SARS-CoV-2 solo mostró reactividad cruzada con el plasma que tenía anticuerpos contra SARS-CoV, pero no hubo reactividad frente a los otros coronavirus humanos30. Quitando esta situación la prueba demostró alta especificidad. En cuanto a la sensibilidad, IgM mostró 76% de sensibilidad al día 7 desde inicio de síntomas, en casos confirmados por método molecular. La media de aparición de IgM fue el día 5 desde inicio de los síntomas con un intervalo intercuartílico de 3-6 días. La IgG mostró una sensibilidad de 78%. La media de aparición de IgG fue el día 14 desde el inicio de los síntomas con un intervalo intercuartílico de 10-18 días. En el estudio de Zhao J y colaboradores32 la media de aparición de IgM fue el día 12 y mostró una sensibilidad de 82,7%, con gran variación en los días de aparición de respuesta. En el caso de IgG la media de aparición fue el día 14 con una sensibilidad de 64,7%.
Tipos de pruebas serológicas comercializadas
En cuanto a los tipos de pruebas comercializados, hasta el momento, a nivel mundial, se han comercializado tres tipos de pruebas.
- Ensayos de inmunoabsorción ligado a enzimas (ELISA).
- Pruebas rápidas que detectan IgM/IgG en soporte inmunocromatográfico.
- Ensayos automatizados por quimioluminiscencia.
Hasta el 24 de junio de 2020, la US Food and Drug Administration (FDA) ha emitido autorizaciones de uso de emergencia (EUA) a varias pruebas serológicas. Mayoritariamente se trata de ensayos automatizados por quimioluminiscencia. Pero también ensayos de ELISA y pruebas rápidas que detectan IgM e IgG33. EUA implica que la empresa comercial ha enviado a FDA información sobre el desempeño del ensayo para su revisión y aprobación.
El FIND, a través de varias instituciones colaboradoras, evaluó varios ensayos rápidos de detección de IgM/IgG obteniendo resultados muy variables en relación tanto a sensibilidad como especificidad34.
Dados estos hallazgos se hace necesario evaluar los ensayos a comercializar en Uruguay para conocer su desempeño analítico y clínico.
Si contáramos con ensayos serológicos con un buen desempeño, serían de utilidad cuando los pacientes consultan con complicaciones tardías de la enfermedad, en casos de RT-PCR falsamente negativa, dado que la eliminación viral disminuye con el tiempo8.
Por otra parte, ensayos serológicos que evalúen con precisión la infección previa y la inmunidad al SARS-CoV-2 serán esenciales para los estudios epidemiológicos, la vigilancia continua, los estudios de vacunas y potencialmente para la evaluación del riesgo de los trabajadores de la salud8,35.
Conclusiones
Al día de hoy el diagnóstico de COVID-19 se debe realizar a través de métodos moleculares, vigilando el cumplimiento de la etapa preanalítica con la finalidad de minimizar los falsos negativos. Las técnicas que detectan antígenos son prometedoras, pero falta evaluación local. La pruebas serológicas que lograran un desempeño satisfactorio serán de utilidad para pacientes con síntomas respiratorios pero que no consultan en la fase inicial o aguda. Y, en un futuro, van a ser fundamentales para conocer el estatus inmunitario de la población.